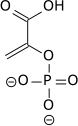
Enolaza


Enolaza (EC 4.2.1.11) jest enzymem z klasy liaz. Przeprowadza przedostatnią reakcję w glikolizie, katalizując dehydratację 2-fosfoglicerynianu (2PG) do fosfoenolopirogronianu (PEP).
Struktura[2]
[edytuj | edytuj kod]W budowie enolazy można wyróżnić jedenaście motywów harmonijek beta oraz dwanaście helis alfa. Trzy harmonijki beta są zlokalizowane na N-końcu enzymu, tak samo jak cztery alfa helisy. Pozostałe harmonijki beta i alfa helisy formują się na kształt beczułki/cylindra, gdzie zlokalizowane jest centrum aktywne.
Miejsce aktywne zlokalizowane jest w regionach C-końca cylindra, gdzie znajdują się specyficzne miejsca przyłączania kofaktorów, w postaci jonów metalu, oraz substratów, takich jak 2-fosfoglicerynian albo fosfoenolopirogronian.
Reakcja[3]
[edytuj | edytuj kod]Mechanizm[4]
[edytuj | edytuj kod]
Asp246, Glu295 oraz Asp320 utrzymują i stabilizują kation metalu. Przyłączenie kationu metalu (Mg2+) powoduje zmiany konformacyjne, które aktywują enzym i umożliwiają powstanie kompleksu między 2-fosfoglicerynianem a enzymem. Gln167, Arg374 i Lys396 neutralizują i stabilizują negatywnie naładowaną grupę karboksylową 2-fosfoglicerynianu.
Po "zakleszczeniu się" 2-fosfoglicerynianu w miejscu aktywnym, jego grupa fosforanowa łączy się, za pomocą wiązania wodorowego, z pierścieniem imidazolowym His159. Pętle eznymu, które tworzą reszty Val153-Phe169 oraz Ser250-Glu277, przesuwają się w skoordynowany sposób, umożliwiając His159 lepsze ułożenie względem grupy fosforanowej substratu w celu donacji protonu. Negatywny ładunek grupy fosforylowej jest dalej neutralizowany przez Arg374 oraz jon metalu.
Lys345 przysuwa się bliżej Arg374, powodując, że traci sama proton. W konsekwencji, Lys345 zabiera proton pochodzący z C-2 2-fosfoglicerynianu. Ostatecznie, proton (połączony wiązaniem wodorowym z grupą karboksylową 2-fosfoglicerynianu), który dzieliły jednocześnie Glu168 and Glu211 "przeskakuje" na grupę hydroksylową 2-PGA, która rozpada się i formuje wodę. Powstaje podwójne wiązanie między C-2 oraz C-3. Formuje się produkt - fosfoenolopirogronian.
Przypisy
[edytuj | edytuj kod]- ↑ Zhang E, Brewer JM, Minor W, Carreira LA, Lebioda L. Mechanism of enolase: the crystal structure of asymmetric dimer enolase-2-phospho-D-glycerate/enolase-phosphoenolpyruvate at 2.0 A resolution. „Biochemistry”. 36 (41), s. 12526–34, 1997. DOI: 10.1021/bi9712450. PMID: 9376357.
- ↑ Enolase - Structure 3 [1]
- ↑ REACTION: R00658 [2]
- ↑ Enolase active site mechanism: How enolase works [3]