Énolase
| Énolase 1 | ||
 Homodimère d'énolase de levure complexée avec un mélange à l'équilibre de 2PG et de PEP (PDB 1ONE[1]). | ||
| Caractéristiques générales | ||
|---|---|---|
| Symbole | ENO1 | |
| N° EC | 4.2.1.11 | |
| Homo sapiens | ||
| Locus | 1p36.23 | |
| Masse moléculaire | 47 169 Da[2] | |
| Nombre de résidus | 434 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Énolase 2 (neuronale) | ||
| Caractéristiques générales | ||
| Symbole | ENO2 | |
| N° EC | 4.2.1.11 | |
| Homo sapiens | ||
| Locus | 12p13.31 | |
| Masse moléculaire | 47 269 Da[2] | |
| Nombre de résidus | 434 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Énolase 3 (musculaire) | ||
| Caractéristiques générales | ||
| Symbole | ENO3 | |
| N° EC | 4.2.1.11 | |
| Homo sapiens | ||
| Locus | 17p13.2 | |
| Masse moléculaire | 46 987 Da[2] | |
| Nombre de résidus | 434 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
| Énolase 4 | ||
| Caractéristiques générales | ||
| Symbole | ENO4 | |
| N° EC | 4.2.1.11 | |
| Homo sapiens | ||
| Locus | 10q25.3 | |
| Masse moléculaire | 68 821 Da[2] | |
| Nombre de résidus | 628 acides aminés[2] | |
| Liens accessibles depuis GeneCards et HUGO. | ||
L'énolase, également connue sous les noms de phosphopyruvate hydratase ou 2-phosphoglycérate déshydratase, est une métalloenzyme responsable de la catalyse qui convertit le 2-phospho-D-glycérate (2PG) en phosphoénolpyruvate (PEP), la 9e et avant-dernière étape de la glycolyse :

|
H2O + | 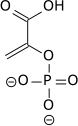
|
| 2-phosphoglycérate | Phosphoénolpyruvate |
L'énolase appartient à la classe de lyases. L'énolase peut aussi catalyser la réaction inverse, selon les concentrations du substrats dans le milieu[3]. Le pH optimal pour cette enzyme est de 6,5[4]. L'énolase est présente dans tous les tissus et organismes capables de réaliser la glycolyse ou la fermentation. L'enzyme a été découverte par Lohmann et Meyerhof en 1934[5] et a depuis été isolée au sein de divers organes sources tels que le muscle humain et les érythrocytes[4].
| N° EC | EC |
|---|---|
| N° CAS | |
| Cofacteur(s) | Magnésium |
| IUBMB | Entrée IUBMB |
|---|---|
| IntEnz | Vue IntEnz |
| BRENDA | Entrée BRENDA |
| KEGG | Entrée KEGG |
| MetaCyc | Voie métabolique |
| PRIAM | Profil |
| PDB | RCSB PDB PDBe PDBj PDBsum |
| GO | AmiGO / EGO |
Isoenzymes
[modifier | modifier le code]L'énolase possède trois sous-unités, α, ß et γ, chacune codée par un gène différent qui peut se combiner pour former cinq isoenzymes différentes : αα, αß, αγ, ββ et γγ[3],[6]. Trois de ces isoenzymes (toutes homodimères) se retrouvent plus fréquemment dans les cellules de l'homme adulte que les autres :
- αα ou énolase non-neuronale (NNE), se retrouve dans toutes sortes de tissus tels que le foie, le cerveau, le rein, la rate
et les tissus adipeux. Elle est également appelée énolase 1 - ββ ou énolase spécifique du muscle (MSE). Connu aussi sous le nom d'énolase 3.
- γγ ou énolase spécifique des neurones (NSE). Également appelée énolase 2.
Structure
[modifier | modifier le code]L'énolase a un poids moléculaire de 82 000 à 100 000 daltons selon son isoforme[3],[4]. Dans l'alpha énolase humaine, les deux sous-unités sont orientées de façon antiparallèle de sorte que le Glu20 d'une sous-unité forme une liaison ionique avec l'Arg414 de l'autre sous-unité[3]. Chaque sous-unité a deux domaines distincts. Le petit domaine N-terminal consiste en trois hélices alpha et quatre feuillets béta[3],[6]. Le grand domaine C-terminal commence par deux feuillets ß suivis par deux hélices α et se termine par un creux entouré de feuillets ß s'alternant avec des hélices α arrangées de façon qu'elles entourent les feuillets ß[3],[6]. La structure compacte et globulaire de l'enzyme résulte d'interactions hydrophobes significatives entre ces deux domaines.
L'énolase est une enzyme très bien gardée avec cinq sites actifs particulièrement importants pour l'activité. Quand on la compare à l'énolase de type sauvage - énolase mutée qui diffère au Glu168, Glu211, Lys345, ou au résidu Lys396 - a un niveau d'activité qui est diminué d'un facteur 105[3]. De la même manière, les changements affectant His159 laissent l'énolase mutée avec seulement 0.01 % de son activité catalytique[3]. Une fonction essentielle de l'énolase est régulée par ses deux cofacteurs Mg2+ dans le site actif, qui servent à stabiliser les charges négatives du substrat[3],[6].
-
Structure 3D d'un dimère d'énolase en orientation antiparallèle. Le Glu20 N-terminal de l'un des dimères forme une liaison ionique avec le Arg414 C-terminal de l'autre pour stabiliser la strucutre quaternaire de l'enzyme.
-
Site actif de l'énolase.
Mécanisme
[modifier | modifier le code]
En utilisant des sondes isotopiques, le mécanisme complet pour la conversion du 2-PG en PEP est une réaction d'élimination du type E1cb impliquant un intermédiaire carbanion[7]. Le mécanisme détaillé qui suit est basé sur des études cristallographie et de cinétique chimique[3],[8],[9],[10],[11],[12],[13]. Quand le substrat, le 2-phosphoglycérate, se lie à l'α-énolase, son groupe carboxyle forme une liaison de coordination avec les deux ions magnésium dans le site actif. Ceci stabilise la charge négative de l'oxygène déprotoné tout en augmentant l'acidité de l'hydrogène alpha. La Lys345 de l'énolase déprotone l'hydrogène alpha et la charge négative résultante est stabilisée sur l'oxygène du carboxylate par la résonance de l'ion cofacteur magnésium. Après la formation de l'intermédiaire carbanion, l'hydroxyde en C3 est éliminé avec l'eau grâce au Glu211, et le PEP est ainsi formé.
De plus, des changements conformationnels aidant la catalyse peuvent survenir au sein de l'enzyme. Dans α-énolase de l'homme, le substrat est orienté dans la position favorisant l'attache de l'enzyme en raison des interactions avec les deux ions catalytiques de magnésium, Gln167 et Lys396. Les mouvements en boucles de la Ser36 vers l'His43, de la Ser158 vers la Gly162 et de l'Asp255 vers l'Asn256 permettent à la Ser39 de se lier au Mg2+ et ainsi enfermer le site actif. En plus de cette coordination avec les ions catalytiques de magnésium, le pKa de l'hydrogène alpha du substrat diminue en raison de la protonation du groupe phosphoryle par l'His159 et de sa proximité avec l'Arg374. L'Arg374 est aussi responsable de la déprotonation de la Lys345 dans le site actif lui fournissant ainsi son rôle primordial dans le mécanisme.
Utilisations diagnostiques
[modifier | modifier le code]Dans des expériences médicales récentes, des concentrations d'énolase ont été échantillonnées dans le but de diagnostiquer certaines affections néoplasiques et leur gravité. Par exemple, de plus fortes concentrations en énolase dans le liquide céphalorachidien (LCR) sont davantage corrélées à la formation d'astrocytome malins qu'ont pu le montrer d'autres enzymes testées (aldolase, pyruvate kinase, créatine kinase et lactate déshydrogénase)[14]. La même étude a montré qu'une évolution plus rapide de la tumeur est apparue chez des patients dont la concentration d'énolase dans le fluide céphalorachidien était plus élevée. Des niveaux accrus d'énolase ont aussi été identifiés chez des patients qui ont souffert récemment d'un infarctus du myocarde ou d'un accident cérébro-vasculaire. On a déduit que les niveaux d'énolase du LCR, de sérum NSE (type γγ) et de créatine kinase (type ßß) sont indicateurs pour l'évaluation de pronostics chez les victimes d'arrêt cardiaque. D'autres études se sont concentrées sur la valeur pronostique des quantités de NSE chez les victimes d'accident cérébro-vasculaires[15]. D'autres études se sont concentrées sur la valeur pronostique des concentrations en NSE chez les victimes d'accident cérébro-vasculaires[16].
Les auto-anticorps dirigés contre nos alpha-énolases sont associés à la maladie rare qu'est l'encéphalopathie d'Hashimoto[17].
Inhibition par le fluorure
[modifier | modifier le code]Le fluorure est un compétiteur bien connu pour le substrat 2-PG qui doit s'attacher à l'énolase. Le fluorure fait partie d'un complexe avec le magnésium et le phosphate, qui se lie au site actif de l'énolase au lieu du 2-PG[4]. De cette manière, boire de l'eau enrichie en fluor fournit du fluorure à un niveau qui empêche l'activité des bactéries énolases de la bouche sans nuire à la santé de la personne. Perturber la voie glycolytique des bactéries - et, ainsi, son fonctionnement métabolique normal - empêche la formation de caries dentaires[18],[19].
Notes et références
[modifier | modifier le code]- (en) Todd M. Larsen, Joseph E. Wedekind, Ivan Rayment et George H. Reed, « A Carboxylate Oxygen of the Substrate Bridges the Magnesium Ions at the Active Site of Enolase: Structure of the Yeast Enzyme Complexed with the Equilibrium Mixture of 2-Phosphoglycerate and Phosphoenolpyruvate at 1.8 Å Resolution », Biochemistry, vol. 35, no 14, , p. 4349-4358 (PMID 8605183, DOI 10.1021/bi952859c, lire en ligne)
- Les valeurs de la masse et du nombre de résidus indiquées ici sont celles du précurseur protéique issu de la traduction du gène, avant modifications post-traductionnelles, et peuvent différer significativement des valeurs correspondantes pour la protéine fonctionnelle.
- (en) Pancholi V, « Multifunctional α-enolase: its role in diseases », Cell Mol Life Sci., vol. 58, no 7, , p. 902–20 (PMID 11497239, DOI 10.1007/PL00000910, lire en ligne)
- (en) Hoorn RK, Flickweert JP, Staal GE, « Purification and properties of enolase of human erythroctyes », Int J Biochem, vol. 5, nos 11–12, , p. 845–52 (DOI 10.1016/0020-711X(74)90119-0)
- (de) Lohman K & Meyerhof O (1934), Über die enzymatische umwandlung von phosphoglyzerinsäure in brenztraubensäure und phosphorsäure (Transformation enzymatique de l'acide phosphoglycérique en acides pyruvique et phosphorique). Biochem Z 273, 60–72.
- (en) Peshavaria M, Day IN, « Molecular structure of the human muscle-specific enolase gene (ENO3) », Biochem J., vol. 275 (Pt 2), no Pt 2, , p. 427–33 (PMID 1840492, PMCID 1150071)
- (en) Dinovo EC, Boyer PD, « Isotopic probes of the enolase reaction mechanism », J Biol Chem, vol. 240, , p. 4586–93
- (en) Poyner RR, Laughlin LT, Sowa GA, Reed GH, « Toward identification of acid/base catalysts in the active site of enolase: comparison of the properties of K345A, E168Q, and E211Q variants », Biochemistry, vol. 35, no 5, , p. 1692–9 (PMID 8634301, DOI 10.1021/bi952186y)
- (en) Reed GH, Poyner RR, Larsen TM, Wedekind JE, Rayment I, « Structural and mechanistic studies of enolase », Curr Opin Struct Biol., vol. 6, no 6, , p. 736–43 (PMID 8994873, DOI 10.1016/S0959-440X(96)80002-9, lire en ligne)
- (en) Wedekind JE, Reed GH, Rayment I, « Octahedral coordination at the high-affinity metal site in enolase: crystallographic analysis of the MgII—enzyme complex from yeast at 1.9 Å resolution », Biochemistry, vol. 34, no 13, , p. 4325–30 (PMID 7703246, DOI 10.1021/bi00013a022)
- (en) Wedekind JE, Poyner RR, Reed GH, Rayment I, « Chelation of serine 39 to Mg2+ latches a gate at the active site of enolase: structure of the bis(Mg2+) complex of yeast enolase and the intermediate analog phosphonoacetohydroxamate at 2.1-Å resolution », Biochemistry, vol. 33, no 31, , p. 9333–42 (PMID 8049235, DOI 10.1021/bi00197a038)
- (en) Larsen TM, Wedekind JE, Rayment I, Reed GH, « A carboxylate oxygen of the substrate bridges the magnesium ions at the active site of enolase: structure of the yeast enzyme complexed with the equilibrium mixture of 2-phosphoglycerate and phosphoenolpyruvate at 1.8 Å resolution », Biochemistry, vol. 35, no 14, , p. 4349–58 (PMID 8605183, DOI 10.1021/bi952859c)
- (en) Duquerroy S, Camus C, Janin J, « X-ray structure and catalytic mechanism of lobster enolase », Biochemistry, vol. 34, no 39, , p. 12513–23 (PMID 7547999, DOI 10.1021/bi00039a005)
- (en) Royds JA, Timperley WR, Taylor CB, « Levels of enolase and other enzymes in the cerebrospinal fluid as indices of pathological change », J Neurol Neurosurg Psychiatr., vol. 44, no 12, , p. 1129–35 (PMID 7334408, PMCID 491233, DOI 10.1136/jnnp.44.12.1129)
- (en) Roine RO, Somer H, Kaste M, Viinikka L, Karonen SL, « Neurological outcome after out-of-hospital cardiac arrest. Prediction by cerebrospinal fluid enzyme analysis », Arch Neurol., vol. 46, no 7, , p. 753–6 (PMID 2742544)
- (en) Hay E, Royds JA, Davies-Jones GA, Lewtas NA, Timperley WR, Taylor CB, « Cerebrospinal fluid enolase in stroke », J Neurol Neurosurg Psychiatr., vol. 47, no 7, , p. 724–9 (PMID 6747647, PMCID 1027902, DOI 10.1136/jnnp.47.7.724)
- (en) Fujii A, Yoneda M, Ito T, Yamamura O, Satomi S, Higa H, Kimura A, Suzuki M, Yamashita M, Yuasa T, Suzuki H, Kuriyama M, « Autoantibodies against the amino terminal of alpha-enolase are a useful diagnostic marker of Hashimoto's encephalopathy », J. Neuroimmunol., vol. 162, nos 1–2, , p. 130–6 (PMID 15833368, DOI 10.1016/j.jneuroim.2005.02.004)
- (en) Centers For Disease Control, « Populations receiving optimally fluoridated public drinking water—United States, 2000 », MMWR Morb Mortal Wkly Rep., vol. 51, no 7, , p. 144–7 (PMID 11905481)
- (en) Hüther FJ, Psarros N, Duschner H, « Isolation, characterization, and inhibition kinetics of enolase from Streptococcus rattus FA-1 », Infect Immun., vol. 58, no 4, , p. 1043–7 (PMID 2318530, PMCID 258580, lire en ligne)
- (en) Cet article est partiellement ou en totalité issu de l’article de Wikipédia en anglais intitulé « Enolase » (voir la liste des auteurs).




