Código xenético
O código xenético é o conxunto de normas polas que a información codificada no material xenético (secuencias de ADN ou ARN) se traduce a proteínas (secuencias de aminoácidos) nas células vivas. O código define as correspondencias entre as distintas secuencias de tres nucleótidos, chamadas codóns, e aminoácidos. Un codón corresponde a un aminoácido específico.
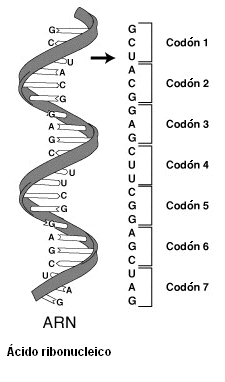

A secuencia do material xenético componse de catro bases nitroxenadas distintas, que teñen unha función equivalente a letras no código xenético: adenina (A), timina (T), guanina (G) e citosina (C) no ADN e adenina (A), uracilo (U), guanina (G) e citosina (C) no ARN.
Se agrupamos de tres en tres os catro nucleótidos que hai podendo repetilos obtemos 64 combinacións distintas posibles, que son os 64 codóns do código xenético. Deles, 61 codifican aminoácidos (un deles, AUG, é ademais o codón de inicio) e os tres restantes son codóns de parada (UAA, chamado ocre; UAG, denominado ámbar; UGA ou ópalo). A secuencia de codóns determina a secuencia aminoacídica dunha proteína concreta, que terá unha estrutura e unha función específicas.
Descubrimento do código xenético
editarCando James Watson, Francis Crick, Maurice Wilkins e Rosalind Franklin descubriron a estrutura do ADN, empezou a estudarse en profundidade o proceso de tradución das proteínas. En 1955, Severo Ochoa e Marianne Grunberg-Manago illaron o encima polinucleótido fosforilase, que tiña a capacidade de sintetizar ARNm a partir de calquera tipo de nucleótidos que houbera no medio sen precisar dun modelo. Así, a partir dun medio no cal había só UDP (uridín difosfato) sintetizábase un ARNm que constaba unicamente dunha repetición de ácido 5'-uridílico, o denominado poli-U (....UUUUU....). George Gamow postulou que as células debían empregar un código de codóns de tres bases para codificar a secuencia aminoacídica, xa que tres é o número enteiro mínimo que con catro bases nitroxenadas distintas permite máis de 20 combinacións (64 exactamente).
Que os codóns constan de tres nucleótidos foi demostrado por primeira vez no experimento de Crick, Brenner e colaboradores. Marshall Nirenberg e Heinrich J. Matthaei en 1961 nos Institutos Nacionais da Saúde descubriron a primeira correspondencia codón-aminoácido. Empregando un sistema libre de células, traduciron unha secuencia ARN de poli-uracilo (UUU...) e descubriron que o polipéptido que sintetizaran só contiña fenilalanina. Disto dedúcese que o codón UUU específica o aminoácido fenilalanina. Continuando co traballo anterior, Nirenberg e Philip Leder puideron determinar a tradución de 54 codóns, utilizando diversas combinacións de ARNm, pasadas a través dun filtro que contiña ribosomas. Os ARNt uníanse a tripletes específicos.
Posteriormente, Har Gobind Khorana completou o código, e pouco despois, Robert W. Holley determinou a estrutura do ARN de transferencia, a molécula adaptadora que facilita a tradución. Este traballo baseouse en estudos anteriores de Severo Ochoa, quen recibiu o premio Nobel en 1959 polo seu traballo na encimoloxía da síntese de ARN. En 1968, Khorana, Holley e Nirenberg recibiron o Premio Nobel de Fisioloxía e Medicina polo seu traballo.
Transferencia de información
editarO xenoma dun organismo encóntrase no ADN ou, no caso dalgúns virus, no ARN. A porción de xenoma que codifica unha (ou varias) proteínas ou un ARN coñécese como xene. Os xenes que codifican proteínas están compostos por unidades de trinucleótidos chamadas codóns, cada unha das cales codifica un aminoácido. Cada subunidade nucleotídica está formada por un fosfato, unha desoxirribosa e unha das catro posibles bases nitroxenadas. As bases purínicas adenina (A) e guanina (G) son máis grandes e teñen dous aneis aromáticos. As bases pirimidínicas citosina (C) e timina (T) son máis pequenas e só teñen un anel aromático. Na configuración en dobre hélice, dúas cadeas de ADN están unidas entre si por pontes de hidróxeno nunha asociación coñecida como emparellamento de bases. Ademais, estas pontes sempre se forman entre unha adenina dunha cadea e unha timina da outra e entre unha citosina dunha cadea e unha guanina da outra. Isto quere dicir que o número de residuos A e T será o mesmo nunha dobre hélice e o mesmo pasará co número de residuos de G e C. No ARN, a timina (T) substitúese por uracilo (U), e a desoxirribosa por unha ribosa.
Cada xene codificante para proteína transcríbese nunha molécula que servirá como molde ou patrón, que se coñece como ARN mensaxeiro ou ARNm. Este, á súa vez, tradúcese no ribosoma, orixinando unha cadea aminoacídica ou polipeptídica. No proceso de tradución necesítase un ARN de transferencia ou ARNt específico para cada aminoácido, que leva o aminoácido unido a el covalentemente, guanosina trifosfato como fonte de enerxía e certos factores de tradución. Os ARNt teñen anticodóns complementarios aos codóns do ARNm e pódense “cargar” covalentemente no seu extremo 3' terminal CCA con aminoácidos. Os ARNt individuais cárganse cada un cun aminoácido específico grazas á acción dos encimas chamados aminoacil ARNt sintetases, que teñen alta especificidade tanto polos aminoácidos coma polos ARNt. A alta especificidade destes encimas é o motivo fundamental do mantemento da fidelidade da tradución de proteínas.
Pódense facer 4³ = 64 combinacións diferentes con tripletes de tres nucleótidos, xa que matematicamente ditas combinacións son variacións con repetición. Os 64 codóns están asignados a un aminoácido ou a sinais de parada na tradución. Se, por exemplo, temos unha secuencia de ARN, UUUAAACCC, e a lectura do fragmento empeza no primeiro U (convenio 5' a 3'), habería tres codóns que serían UUU, AAA e CCC, cada un dos cales específica un aminoácido. Esta secuencia de ARN traducirase nunha secuencia aminoacídica de tres aminoácidos de lonxitude. Pódese comparar coa informática, onde un codón se asemellaría a unha palabra, o que sería a peza estándar para o manexo de datos (como o é un aminoácido para unha proteína), e un nucleótido é similar a un bit, que sería a unidade máis pequena. (Na práctica, precisariamos polo menos 2 bits para representar un nucleótido, e 6 para un codón, nun computador normal).
Características
editarUniversalidade
editarO código xenético é compartido por todos os organismos coñecidos, incluíndo virus e orgánulos, aínda que poden aparecer pequenas diferenzas. Así, por exemplo, o codón UUU codifica o aminoácido fenilalanina tanto en bacterias, coma en arqueas e en eucariontes. Este feito indica que o código xenético tivo unha orixe única en todos os seres vivos.
Grazas á xenética molecular, identificáronse 22 códigos xenéticos, que se diferencian do chamado código xenético estándar polo significado dun ou máis codóns. A maior diversidade preséntase nas mitocondrias, orgánulos das células eucariotas que se orixinaron evolutivamente a partir de Bacteria a través dun proceso de endosimbiose. O código do xenoma nuclear dos eucariotas, cando ten diferenzas co código estándar, só adoita diferenciarse nos codóns de iniciación e terminación.
Especificidade e continuidade
editarNingún codón codifica máis dun aminoácido, xa que, de non ser así, isto comportaría problemas considerables para a síntese de proteínas específicas para cada xene. Tampouco presenta solapamento: os tripletes están dispostos de maneira liñal e continua, de maneira que entre eles non existan comas nin espazos, e os tripletes non comparten ningunha base nitroxenada. A súa lectura faise nun só sentido (5' → 3'), desde o codón de iniciación ata o codón de parada. Porén, nun mesmo ARNm poden existir varios codóns de inicio, o que orixina a síntese de varios polipéptidos diferentes a partir do mesmo transcrito.
Dexeneración
editarO código xenético ten redundancia pero non ambigüidade (ver táboas de codóns). Por exemplo, aínda que os codóns GAA e GAG especifican os dous o ácido glutámico (redundancia), ningún específica outro aminoácido (non hai ambigüidade). Os codóns que codifican un aminoácido poden diferir nalgunha das súas tres posicións, por exemplo, o ácido glutámico especfícase por GAA e GAG (difiren na terceira posición), o aminoácido leucina especifícase polos codóns UUA, UUG, CUU, CUC, CUA e CUG (difiren na primeira ou na terceira posición), entanto que no caso da serina, especifícase por UCA, UCG, UCC, UCU, AGU, AGC (difiren na primeira, segunda ou tercera posición).
Dise que unha posición dun codón é catro veces degenerada se con calquera nucleótido nesta posición se especifica o mesmo aminoácido. Por exemplo, a terceira posición dos codóns da glicina (GGA, GGG, GGC, GGU) é catro veces dexenerada, porque todas as substitucións de nucleótidos nese lugar son sinónimos; é dicir, non varían o aminoácido. Só a terceira posición dalgúns codóns pode ser catro veces dexenerada. Dise que unha posición dun codón é dúas veces dexenerada se só dúas das catro posibles substitucións de nucleótidos especifican o mesmo aminoácido. Por exemplo, a terceira posición dos codóns do ácido glutámico (GAA, GAG) é dobre dexenerada. Nos lugares dúas veces dexenerados, os nucleótidos equivalentes son sempre dúas purinas (A/G) ou dúas pirimidinas (C/U), así que só as chamadas substitucións transversionais (purina a pirimidina ou pirimidina a purina) en dobres dexenerados son antónimas. Dise que unha posición dun codón é non dexenerada se unha mutación nesa posición ten como resultado a substitución dun aminoácido. Só hai un sitio triplo dexenerado no que cambiando tres de catro nucleótidos non hai efecto no aminoácido, mientres que cambiando os catro posibles nucleótidos aparece unha substitución do aminoácido. trátase da terceira posición dun codón de isoleucina: AUU, AUC e AUA, todos codifican isoleucina, pero AUG codifica metionina. En biocomputación, este sitio trátase a miúdo como dobre dexenerado.
Hai tres aminoácidos codificados por 6 codóns diferentes: serina, leucina, arxinina. Só dous aminoácidos se especifican por un único codón; un deles é a metionina, especificada por AUG, que tamén indica o comezo da tradución; o outro é o triptófano, especificado por UGG. Que o código xenético sexa dexenerado é o que determina a posibilidade de mutacións sinónimas.
A dexeneración aparece porque o código xenético designa 20 aminoácidos e mais o sinal de parada. Debido a que hai catro bases, os codóns de tripletes deberán producir polo menos 21 códigos diferentes. Por exemplo, se houbese dúas bases por codón, entón só poderían codificarse 16 aminoácidos (4²=16). E dado que polo menos se necesitan 21 códigos, 4³ dá 64 codóns posibles, indicando que debe haber dexeneración.
Esta propiedade do código xenético faino máis tolerante aos fallos das mutacións puntuais. Por exemplo, en teoría, os codóns catro veces dexenerados poden tolerar calquera mutación puntual na terceira posición, aínda que o codón de uso nesgado restrinxe isto na práctica en moitos organismos; os que son dúas veces dexenerados poden tolerar unha das tres posibles mutacións puntuais na terceira posición. Debido a que as mutacións de transición (purina a purina ou pirimidina a pirimidina) son máis probables que as de transversión (purina a pirimidina ou viceversa), a equivalencia de purinas ou de pirimidinas nos lugares dobres dexenerados engade unha tolerancia complementaria aos fallos.
Agrupamento de codóns por residuos aminoacídicos, volume molar e hidropatía
editarUnha consecuencia práctica da redundancia é que algúns erros do código xenético só causan unha mutación silenciosa ou un erro que non afectará á proteína porque a súa hidrofilidade ou hidrofobidade se mantén por unha substitución equivalente de aminoácidos; por exemplo, un codón de NUN (N =calquera nucleótido) tende a codificar un aminoácido hidrofóbico. NCN codifica residuos aminoacídicos que son pequenos en canto a tamaño e moderados en canto a hidropatía (non son nin fortemente hidrofílicos nin fortemente hidrofóbicos); NAN codifica residuos hidrofílicos dun tamaño medio; UNN codifica residuos que non son hidrofílicos.[1][2] Estas tendencias poden ser resultado dunha relación das aminoacil ARNt sintetases cos codóns herdada dun devanceiro común dos seres vivos.
Mesmo así, as mutacións puntuais poden causar a aparición de proteínas disfuncionais. Por exemplo, un xene de hemoglobina mutado provoca a enfermidade das células falciformes ou anemia falciforme. Na hemoglobina mutante un glutamato hidrofílico (Glu) substitúese por unha valina hidrofóbica (Val), e isto débese a que GAA ou GAG se converteron en GUA ou GUG. A substitución de glutamato por valina reduce a solubilidade de β-globina que provoca que a hemoglobina forme polímeros liñais unidos por interaccións hidrofóbicas entre os grupos de valina e causando a deformación falciforme dos eritrocitos. A doenza das células falciformes non está causada xeralmente por unha mutación de novo. Máis ben selecciónase en rexións onde é frecuente a malaria (de forma parecida á talasemia), xa que os individuos heterocigotos presentan certa resistencia ante o parasito malárico Plasmodium (vantaxe heterocigótica ou heterose).
A relación entre o ARNm e o ARNt a nivel da terceira base pódese producir por bases modificadas na primeira base do anticodón do ARNt, e os pares de bases formados chámanse “pares de bases cambaleantes”. As bases modificadas inclúen inosina e os pares de bases que non son do tipo de Watson e Crick U-G.
Usos incorrectos do termo
editarA expresión "código xenético" é frecuentemente utilizada nos medios de comunicación como sinónimo de xenoma, xenotipo, información xenética ou de ADN. Frases como «Analizouse o código xenético dos restos e coincidiu co da desaparecida», ou «crearase unha base de datos co código xenético de todos os cidadáns» son cientificamente incorrectas tendo en conta o significado de código xenético. Non é correcto falar do «código xenético dunha determinada persoa», porque o código xenético é o mesmo para todos os individuos (e, basicamente, para todas as especies!). Porén, cada organismo ten un xenotipo propio, aínda que é posible que o comparta con outros (caso dos xemelgos univitelinos ou individuos orixinados por reprodución asexual). As distintas especies conteñen diferente información xenética, pero "escribiron" esa información usando o mesmo código xenético. Por tanto, un home, unha ra e un piñeiro terán o mesmo código xenético pero distinta información xenética nos seus xenomas.
Táboa do código xenético estándar
editarO código xenético estándar é o que aparece nas seguintes táboas. A táboa 1 mostra que aminoácido específica cada un dos 64 codóns. A táboa 2 mostra os codóns que especifican cada un dos 20 aminoácidos que interveñen na tradución. Estas táboas chámanse táboas de avance e retroceso respectivamente. Por exemplo, o codón AAU é o aminoácido asparaxina, e UGU e UGC representan cisteína (na denominación estándar de 3 letras, Asn e Cys, respectivamente).
| apolar | polar | básico | ácido | codón de parada |
| 2ª base | |||||
|---|---|---|---|---|---|
| U | C | A | G | ||
| 1ª base |
U | UUU (Phe/F) Fenilalanina UUC (Phe/F) Fenilalanina |
UCU (Ser/S) Serina UCC (Ser/S) Serina |
UAU (Tyr/Y) Tirosina UAC (Tyr/Y) Tirosina |
UGU (Cys/C) Cisteína UGC (Cys/C) Cisteína |
| UUA (Leu/L) Leucina | UCA (Ser/S) Serina | UAA Parada (Ocre) | UGA Parada (Ópalo) | ||
| UUG (Leu/L) Leucina | UCG (Ser/S) Serina | UAG Parada (Ámbar) | UGG (Trp/W) Triptófano | ||
| C | CUU (Leu/L) Leucina CUC (Leu/L) Leucina |
CCU (Pro/P) Prolina CCC (Pro/P) Prolina |
CAU (His/H) Histidina CAC (His/H) Histidina |
CGU (Arg/R) Arxinina CGC (Arg/R) Arxinina | |
| CUA (Leu/L) Leucina CUG (Leu/L) Leucina |
CCA (Pro/P) Prolina CCG (Pro/P) Prolina |
CAA (Gln/Q) Glutamina
CAG (Gln/Q) Glutamina |
CGA (Arg/R) Arxinina CG→G (Arg/R) Arxinina | ||
| A | AUU (Ile/I) Isoleucina AUC (Ile/I) Isoleucina |
ACU (Thr/T) Treonina ACC (Thr/T) Treonina |
AAU (Asn/N) Asparaxina AAC (Asn/N) Asparaxina |
AGU (Ser/S) Serina AGC (Ser/S) Serina | |
| AUA (Ile/I) Isoleucina | ACA (Thr/T) Treonina | AAA (Lys/K) Lisina | AGA (Arg/R) Arxinina | ||
| AUG (Met/M) Metionina, Comienzo | ACG (Thr/T) Treonina | AAG (Lys/K) Lisina | AGG (Arg/R) Arxinina | ||
| G | GUU (Val/V) Valina GUC (Val/V) Valina |
GCU (Ala/A) Alanina GCC (Ala/A) Alanina |
GAU (Asp/D) Ácido aspártico GAC (Asp/D) Ácido aspártico |
GGU (Gly/G) Glicina GGC (Gly/G) Glicina | |
| GUA (Val/V) Valina GUG (Val/V) Valina |
GCA (Ala/A) Alanina GCG (Ala/A) Alanina |
GAA (Glu/E) Ácido glutámico GAG (Glu/E) Ácido glutámico |
GGA (Gly/G) Glicina GGG (Gly/G) Glicina | ||
Nótese que o codón AUG codifica a metionina pero ademais serve de codón de iniciación; o primeiro AUG nun ARNm é a rexión que codifica o sitio onde se inicia a tradución de proteínas.
A seguinte táboa inversa indica os codóns que codifican cada un dos aminoácidos.
| Ala (A) | GCU, GCC, GCA, GCG | Lys (K) | AAA, AAG |
|---|---|---|---|
| Arg (R) | CGU, CGC, CGA, CGG, AGA, AGG | Met (M) | AUG |
| Asn (N) | AAU, AAC | Phe (F) | UUU, UUC |
| Asp (D) | GAU, GAC | Pro (P) | CCU, CCC, CCA, CCG |
| Cys (C) | UGU, UGC | Sec (U) | UGA |
| Gln (Q) | CAA, CAG | Ser (S) | UCU, UCC, UCA, UCG, AGU, AGC |
| Glu (E) | GAA, GAG | Thr (T) | ACU, ACC, ACA, ACG |
| Gly (G) | GGU, GGC, GGA, GGG | Trp (W) | UGG |
| His (H) | CAU, CAC | Tyr (Y) | UAU, UAC |
| Ile (I) | AUU, AUC, AUA | Val (V) | GUU, GUC, GUA, GUG |
| Leu (L) | UUA, UUG, CUU, CUC, CUA, CUG | ||
| Comezo | AUG | Parada | UAG, UGA, UAA |
Aminoácidos 21 e 22
editarExiten outros dous aminoácidos codificados polo código xenético nalgunhas circunstancias e nalgúns organismos. Son a seleniocisteína e a pirrolisina.
A selenocisteína (Sec/U)[3] é un aminoácido presente en multitude de encimas (glutatión peroxidases, tetraiodotironina 5' desiodinases, tiorredoxina redutases, formiato deshidroxenases, glicina redutases e algunhas hidroxenases). Está codificado polo codón UGA (que normalmente é de parada) cando están presentes na secuencia os elementos SECIS (Secuencia de Inserción da Seleniocisteína).
O outro aminoácido, a pirrolisina (Pyl/O),[4][5] é un aminoácido presente en encimas metabólicos de arqueas metanóxenas. Está codificado polo codón UAG (que normalmente é de parada) cando a bacteria ten os xenes PylT e PylS e están presentes na secuencia os elementos PYLIS (Secuencia de Inserción da Pirrolisina).
Excepcións á universalidade
editarComo se mencionou con anterioridade, coñécense 22 códigos xenéticos. Velaquí algunhas diferenzas co estándar:
| Mitocondrias de vertebrados | AGA | Ter | * |
| AGG | Ter | * | |
| AUA | Met | M | |
| UGA | Trp | W | |
| Mitocondrias de invertebrados | AGA | Ser | S |
| AGG | Ser | S | |
| AUA | Met | M | |
| UGA | Trp | W | |
| AGG | Ausente en Drosophila | ||
| Mitocondrias de lévedos | AUA | Met | M |
| CUU | Thr | T | |
| CUC | Thr | T | |
| CUA | Thr | T | |
| CUG | Thr | T | |
| UGA | Trp | W | |
| CGA | Ausente | ||
| CGC | Ausente | ||
| Ciliados, Dasycladaceae e Hexamita (núcleo) | UAA | Gln | Q |
| UAG | Gln | Q | |
| Mitocondrias de balores, protozoos e Coelenterata Mycoplasma e Spiroplasma (núcleo) |
UGA | Trp | W |
| Mitocondrias de equinodermos e platihelmintos | AAA | Asn | N |
| AGA | Ser | S | |
| AGG | Ser | S | |
| UGA | Trp | W | |
| Euplotidae (núcleo) | UGA | Cys | C |
| Endomycetales (núcleo) | CUG | Ser | S |
| Mitocondrias de Ascidiacea | AGA | Gly | G |
| AGG | Gly | G | |
| AUA | Met | M | |
| UGA | Trp | W | |
| Mitocondrias de platihelmintos (alternativo) | AAA | Asn | N |
| AGA | Ser | S | |
| AGG | Ser | S | |
| UAA | Tyr | Y | |
| UGA | Trp | W | |
| Blepharisma (núcleo) | UAG | Gln | Q |
| Mitocondrias de Chlorophyceae | TAG | Leu | L |
| Mitocondrias de trematodos | TGA | Trp | W |
| ATA | Met | M | |
| AGA | Ser | S | |
| AGG | Ser | S | |
| AAA | Asn | N | |
| Mitocondrias de Scenedesmus obliquus | TCA | Ter | * |
| TAG | Leu | L | |
A orixe do código xenético
editarMalia as variacións que existen, os códigos xenéticos utilizados por todas as formas coñecidas de vida son moi similares. Isto suxire que o código xenético se estableceu moi cedo na historia da vida e que ten unha orixe común para as formas de vida actuais. Análises filoxenéticas suxiren que as moléculas de ARNt evolucionaron antes que o actual conxunto de aminoacil-ARNt sintetases.[6]
O código xenético non é unha asignación aleatoria dos codóns a aminoácidos.[7] Por exemplo, os aminoácidos que comparten a mesma vía biosintética tenden a ter a primeira base igual nos seus codóns[8] e aminoácidos con propiedades físicas similares tenden a ter similares codóns.[9][10]
Experimentos recentes demostran que algúns aminoácidos teñen afinidade química selectiva polos seus codóns.[11] Isto suxire que o complexo mecanismo actual de tradución do ARNm que implica a acción de ARNt e encimas asociados, pode ser un desenvolvemento posterior e que, ao primeiro, as proteínas se sintetizasen directamente sobre a secuencia de ARN, actuando este como ribozima e catalizando a formación de enlaces peptídicos (tal como ocorre co ARNr 23S do ribosoma).
Presentouse tamén a hipótese de que o código xenético estándar actual xurdiu por expansión biosintética dun código simple anterior. A vida primordial puido adicionar novos aminoácidos (por exemplo, subprodutos do metabolismo), algúns dos cales se incorporaron máis tarde á maquinaria de codificación xenética. Téñense probas, aínda que circunstanciais, de que formas de vida primitivas empregaban un menor número de aminoácidos diferentes,[12] pero non se sabe con exactitude que aminoácidos e en que orde entraron no código xenético.
Outro factor interesante a termos en conta é que a selección natural favoreceu a dexeneración do código para minimizar os efectos das mutacións[13] . Isto levou a pensar que o código xenético primitivo puido constar de codóns de dous nucleótidos, o que é bastante coherente coa hipóteses do balanceo do ARNt durante o seu acoplamiento (a terceira base non establece pontes de hidróxeno de Watson e Crick).
Notas
editar- ↑ Yang et al. 1990. In Reaction Centers of Photosynthetic Bacteria. M.-E. Michel-Beyerle. (Ed.) (Springer-Verlag, Germany) 209-218
- ↑ Genetic Algorithms and Recursive Ensemble Mutagenesis in Protein Engineering http://www.complexity.org.au/ci/vol01/fullen01/html/ Arquivado 15 de marzo de 2011 en Wayback Machine.
- ↑ IUPAC-IUBMB Joint Commission on Biochemical Nomenclature (JCBN) and Nomenclature Committee of IUBMB (NC-IUBMB) (1999). "Newsletter 1999". European Journal of Biochemistry 264 (2): 607–609. doi:10.1046/j.1432-1327.1999.news99.x. Arquivado dende o orixinal (reprint, with permission) o 09 de xuño de 2011. Consultado o 19 de xuño de 2011.
- ↑ John F. Atkins and Ray Gesteland (2002). "The 22nd Amino Acid. Science 296 (5572): 1409–1410.".
- ↑ Krzycki J (2005). "The direct genetic encoding of pyrrolysine. Curr Opin Microbiol 8 (6): 706–712.".
- ↑ De Pouplana, L.R.; Turner, R.J.; Steer, B.A.; Schimmel, P. (1998). "Genetic code origins: tRNAs older than their synthetases?". Proceedings of the National Academy of Sciences 95 (19): 11295. PMID 9736730. doi:10.1073/pnas.95.19.11295. Arquivado dende o orixinal o 09 de marzo de 2006. Consultado o 19 de xuño de 2011.
- ↑ Freeland SJ, Hurst LD (1998). "The genetic code is one in a million". J. Mol. Evol. 47 (3): 238–48. PMID 9732450. doi:10.1007/PL00006381. Arquivado dende o orixinal o 15 de setembro de 2000. Consultado o 19 de xuño de 2011.
- ↑ Taylor FJ, Coates D (1989). "The code within the codons". BioSystems 22 (3): 177–87. PMID 2650752. doi:10.1016/0303-2647(89)90059-2.
- ↑ Di Giulio M (1989). "The extension reached by the minimization of the polarity distances during the evolution of the genetic code". J. Mol. Evol. 29 (4): 288–93. PMID 2514270. doi:10.1007/BF02103616.
- ↑ Wong JT (1980). "Role of minimization of chemical distances between amino acids in the evolution of the genetic code". Proc. Natl. Acad. Sci. U.S.A. 77 (2): 1083–6. PMID 6928661. doi:10.1073/pnas.77.2.1083.
- ↑ Knight, R.D. and Landweber, L.F. (1998). Rhyme or reason: RNA-arginine interactions and the genetic code. Chemistry & Biology 5(9), R215-R220. PDF version of manuscript Arquivado 12 de maio de 2005 en Wayback Machine.
- ↑ Brooks, Dawn J.; Fresco, Jacques R.; Lesk, Arthur M.; and Singh, Mona. (2002). Evolution of Amino Acid Frequencies in Proteins Over Deep Time: Inferred Order of Introduction of Amino Acids into the Genetic Code Arquivado 13/12/2004, en Wayback Machine.. Molecular Biology and Evolution 19, 1645-1655.
- ↑ Freeland S.J.; Wu T. and Keulmann N. (2003) The Case for an Error Minimizing Genetic Code. Orig Life Evol Biosph. 33(4-5), 457-77.
Véxase tamén
editarOutros artigos
editarLigazóns externas
editar- Servizos online para converter ADN en proteína
- DNA to Amino Acid Conversion
- DNA Sequence -> Protein Sequence converter
- DNA to protein translation (6 frames/17 genetic codes)
- Táboas do código xenético
- Revisións
- Niremberg y Khorana, los hackers del ADN, no Museo Virtual Interactivo sobre a Xenética e o ADN.