Halobacterium
| Halobacterium | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
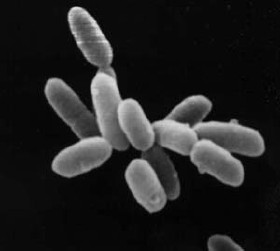 Halobacterium sp. cepa NRC-1, cada célula mide uns 5 μm de lonxitude. | |||||||||||||||
| Clasificación científica | |||||||||||||||
| |||||||||||||||
| Nome binomial | |||||||||||||||
| 'Halobacterium' Elazari-Volcani 1957 | |||||||||||||||
| Especies | |||||||||||||||
| Sinonimia | |||||||||||||||
| |||||||||||||||
Halobacterium é un xénero de arqueas da familia Halobacteriaceae.[1] Como o nome do xénero se estableceu antes da división de bacterias e arqueas en dominios distintos, o nome do xénero acaba en -bacterium, aínda que se trata dun xénero de arqueas.
As especies deste xénero teñen metabolismo aerobio e requiren unha alta concentración de sal no seu ambiente; moitas das súas proteínas non poden funcionar en ambientes con baixas concentracións salinas. Crecen con aminoácidos en aerobiose. As paredes celulares destas bacterias son bastante diferentes das que presentan as bacterias, xa que as membranas lipoproteicas ordinarias non funcionan con altas concentracións salinas. Poden ter forma de bacilos ou de cocos, de cor vermella ou púrpura. Reprodúcense por fisión binaria, e son móbiles. Halobacterium crece mellor a 42 °C. O xenoma de Halobacterium sp. NCR-1 foi secuenciado e está formado por 2 571 010 pares de bases e consta de tres moléculas de ADN circulares, que son: un gran cromosoma e dous pequenos plásmidos. A especie Halobacterium sp. NRC-1, foi moi utilizada nas análises postxenómicas. As especies de Halobacterium poden encontrarse en lagos salgados como o Gran Lago Salgado, o Mar Morto, o Lago Magadi, e calquera outro lago ou lagoa con alta concentración de sal. As especies púrpuras de Halobacterium deben a súa cor á bacteriorrodopsina, unha proteína sensible á luz que lle proporciona enerxía química á célula usando a luz do sol para bombear protóns fóra da célula. O gradiente de protóns resultante a través da membrana da célula utilízase para impulsar a síntese da molécula enerxética ATP. Así, cando estes protóns flúen cara ao interior, son utilizados para a síntese de ATP (este fluxo protónico pode ser emulado cunha diminución do pH fóra da célula, que cause un fluxo de ións H+). A proteína bacteriorrodopsina é quimicamente moi similar ao pigmento detector de luz rodopsina, que se encontra na retina dos vertebrados.
Especies de Halobacterium
[editar | editar a fonte]- Halobacterium cutirubrum > Halobacterium salinarum
- Halobacterium denitrificans > Haloferax denitrificans
- Halobacterium distributum > Halorubrum distributum
- Halobacterium halobium > Halobacterium salinarum
- Halobacterium jilantaiense
- Halobacterium lacusprofundi > Halorubrum lacusprofundi
- Halobacterium mediterranei > Haloferax mediterranei
- Halobacterium noricense
- Halobacterium pharaonis > Natronomonas pharaonis
- Halobacterium piscisalsi
- Halobacterium saccharovorum > Halorubrum saccharovoru
- Halobacterium salinarum
- Halobacterium sodomense > Halorubrum sodomense
- Halobacterium trapanicum > Halorubrum trapanicum
- Halobacterium vallismortis > Haloarcula vallismortis
- Halobacterium volcanii > Haloferax volcanii
Estrutura do xeoma
[editar | editar a fonte]O xenoma de Halobacterium NRC-1 é de 2 571 010 pares de bases distribuídos en tres replicóns circulares, que son: un cromosoma grande de 2 014 239 pb e dous pequenos replicóns (plásmidos) denominados pNRC100 (de 191.346 pb) e pNRC200 (de 365.425 pb). Aínda que son moito máis pequenos que o cromosoma, estes plásmidos conteñen a maioría das 91 secuencias de inserción e inclúen xenes para a ADN polimerase, sete factores de transcrición, xenes para a captación de potasio e fosfato, e para a división celular. O xenoma ten un alto contido G+C de 67,9% no cromosoma grande e 57,9% e 59,2% nos dous plásmidos. Os 91 elementos de secuencias de inserción constitúen 12 familias, e hai 29 destes elementos no pNRC100, 40 no pNRC200, e 22 no cromosoma grande. Isto explica a plasticidade xenética que se observa en xeral nas Halobacteria. Entre as arqueas as Halobacteria son as que teñen unha maior transferencia lateral de xenes.
Estrutura celular e metabolismo
[editar | editar a fonte]As especies de Halobacterium teñen forma bacilar e están envoltas nunha soa membrana formada por unha bicapa lipídica rodeada por unha capa S formada por unha glicoproteína de superficie. As Halobacteria crecen con aminoácidos en condicións aerobias. Aínda que Halobacterium NRC-1 contén xenes para a degradación de glicosa e xenes que codifican encimas para a oxidación dos ácidos graxos, parece que non os poden utilizar como fonte de enerxía. Aínda que o citoplasma mantén un equilibrio osmótico co seu ambiente hipersalino, a célula mantén unha alta concentración de potasio ao utilizaren moitos transportadores activos.
Moitas especies de Halobacterium posúen orgánulos proteináceos chamados vesículas de gas.
Notas
[editar | editar a fonte]- ↑ Ver a páxina do NCBI sobre Halobacterium. Datos extraídos de "NCBI taxonomy resources". National Center for Biotechnology Information. Consultado o 2014-06-27.
Véxase tamén
[editar | editar a fonte]Revistas científicas
[editar | editar a fonte]- DasSarma, S., B.R. Berquist, J.A. Coker, P. DasSarma, J.A. Müller. 2006. Post-genomics of the model haloarchaeon Halobacterium sp. NRC-1. Saline Systems 2:3.Arquivado 02 de outubro de 2011 en Wayback Machine.
- Judicial, Commission of the International Committee on Systematics of Prokaryotes: (2005). "The nomenclatural types of the orders Acholeplasmatales, Halanaerobiales, Halobacteriales, Methanobacteriales, Methanococcales, Methanomicrobiales, Planctomycetales, Prochlorales, Sulfolobales, Thermococcales, Thermoproteales and Verrucomicrobiales are the genera Acholeplasma, Halanaerobium, Halobacterium, Methanobacterium, Methanococcus, Methanomicrobium, Planctomyces, Prochloron, Sulfolobus, Thermococcus, Thermoproteus and Verrucomicrobium, respectively. Opinion 79". Int. J. Syst. Evol. Microbiol. 55 (Pt 1): 517–518. PMID 15653928. doi:10.1099/ijs.0.63548-0.
- Oren A, Ventosa A (2000). "International Committee on Systematic Bacteriology Subcommittee on the taxonomy of Halobacteriaceae. Minutes of the meetings, 16 August 1999, Sydney, Australia". Int. J. Syst. Evol. Microbiol. 50: 1405–1407. PMID 10843089.
Libros científicos
[editar | editar a fonte]- DasSarma, S. 2004. Genome sequence of an extremely halophilic archaeon, in Microbial Genomes, pp. 383–399, C.M. Fraser, T. Read, e K.E. Nelson (eds.), Humana Press, Inc, Totowa, NJ.
- Lynn Margulis, Karlene V.Schwartz, Five Kingdoms. An Illustrated Guide to the Phyla of Life on Earth (W.H.Freeman, San Francisco, 1982) pp. 36–37
- Gibbons, NE (1974). "Family V. Halobacteriaceae fam. nov.". En RE Buchanan and NE Gibbons, eds. Bergey's Manual of Determinative Bacteriology (8th ed.). Baltimore: The Williams & Wilkins Co.
- Elazari-Volcani, B (1957). "Genus XII. Halobacterium Elazari-Volcani, 1940". En RS Breed, EGD Murray, and NR Smith, eds. Bergey's Manual of Determinative Bacteriology (7th ed.). Baltimore: The Williams & Wilkins Co. pp. 207–212.
- Elazari-Volcani, B (1940). "Studies on the microflora of the Dead Sea". Doctoral dissertation, Hebrew University, Jerusalem: 1–116 and i–xiii.
Bases de datos científicas
[editar | editar a fonte]- Referencias de PubMed para Halobacterium.
- Referencias de PubMed Central para Halobacterium.
- Referencias de Google Scholar para Halobacterium.
Bibliografía
[editar | editar a fonte]- PIJPER A (1953). "Halobacterium halobium". J Gen Microbiol 8 (1): viii. PMID 13035061.
- BAYLEY ST, KUSHNER DJ (1964). "THE RIBOSOMES OF THE EXTREMELY HALOPHILIC BACTERIUM, HALOBACTERIUM CUTIRUBRUM". J Mol Biol 9 (3): 654–69. PMID 14216609. doi:10.1016/S0022-2836(64)80173-X.
- Oesterhelt D, Stoeckenius W (1974). "Isolation of the cell membrane of Halobacterium halobium and its fractionation into red and purple membrane". Methods Enzymol. Methods in Enzymology 31 (Pt A): 667–78. ISBN 978-0-12-181894-4. PMID 4418026. doi:10.1016/0076-6879(74)31072-5.
- Oesterhelt D, Stoeckenius W (1971). "Rhodopsin-like protein from the purple membrane of Halobacterium halobium". Nat New Biol 233 (39): 149–52. Bibcode:1971Natur.233..149.. PMID 4940442. doi:10.1038/233149b0.
- Stoeckenius W, Bogomolni RA (1982). "Bacteriorhodopsin and related pigments of halobacteria". Annu Rev Biochem 51: 587–616. PMID 6287921. doi:10.1146/annurev.bi.51.070182.003103.
- Lozier RH, Bogomolni RA, Stoeckenius W (1975). "Bacteriorhodopsin: a light-driven proton pump in Halobacterium Halobium". Biophys J 15 (9): 955–62. Bibcode:1975BpJ....15..955L. PMC 1334761. PMID 1182271. doi:10.1016/S0006-3495(75)85875-9.
- Ng WV, Kennedy SP, Mahairas GG, Berquist B, Pan M, Shukla HD, Lasky SR, Baliga NS, Thorsson V, Sbrogna J, Swartzell S, Weir D, Hall J, Dahl TA, Welti R, Goo YA, Leithauser B, Keller K, Cruz R, Danson MJ, Hough DW, Maddocks DG, Jablonski PE, Krebs MP, Angevine CM, Dale H, Isenbarger TA, Peck RF, Pohlschroder M, Spudich JL, Jung KW, Alam M, Freitas T, Hou S, Daniels CJ, Dennis PP, Omer AD, Ebhardt H, Lowe TM, Liang P, Riley M, Hood L, DasSarma S (2000). "Genome sequence of Halobacterium species NRC-1". Proc Natl Acad Sci U S A 97 (22): 12176–81. Bibcode:2000PNAS...9712176N. PMC 17314. PMID 11016950. doi:10.1073/pnas.190337797.