Ribosoma
Os ribosomas son complexos de proteínas e ARN encargados de levar a cabo a síntese de proteínas. Para iso, empregan a información xenética que reciben do ADN transcrita en forma de ARN mensaxeiro (ARNm). Só son visibles ao microscopio electrónico, debido ao seu reducido tamaño (uns 30 nanómetros). Con microscopio electrónico aparecen como estruturas arredondadas, densas aos electróns. Con microscopio óptico vese que son os responsables da basofilia que presentan algunhas células. Están presentes en todas as células agás nos espermatozoides. Tradicionalmente considéranse orgánulos que carecen de membrana[1][2] ou son tratados como complexos macromoleculares.
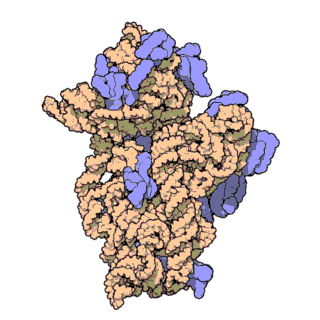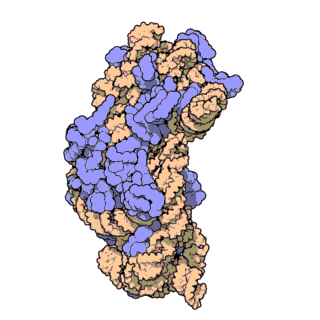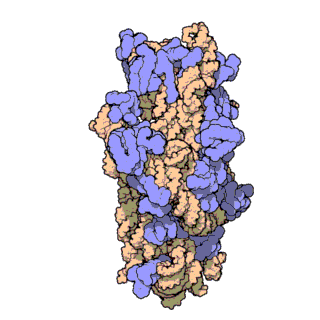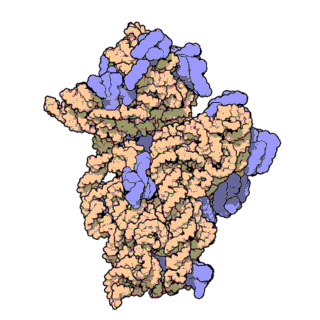
En células eucariotas, os ribosomas fabrícanse no nucléolo do núcleo pero desempeñan a súa función de síntese só no citoplasma. Están formados por ARN ribosómico (ARNr) e por proteínas. Estruturalmente, teñen dúas subunidades de distinto tamaño, maior e menor, formadas por separado no núcleo. Saen do núcleo e poden permanecer separadas no citoplasma, pero para que o ribosoma funcione as dúas subunidades deben estar unidas. Acabada a síntese as subunidades sepáranse de novo. As subunidades mantéñense unidas por cargas eléctricas, e ao diminuír experimentalmente a concentración de Mg2+, as subunidades tenden a separarse. Cando os ribosomas están completos, poden estar illados ou formando grupos (polisomas ou polirribosomas). Tanto os ARNr como as subunidades dos ribosomas poden distinguirse polo seu coeficiente de sedimentación en unidades svedberg (S). En eucariotas os ribosomas do citoplasma son de 80 S. En mitocondrias e plastos de eucariotas, así como en procariotas, son de 70 S. Esta similitude dos ribosomas de bacterias, mitocondrias e plastos indica unha orixe evolutiva común[3][4].
Os exames de cristalografía dos ribosomas mostran que as proteínas do orgánulo non están preto dos sitios de reacción para a síntese dos polipéptidos, o que suxire que os compoñentes proteicos actúan no ribosoma como un armazón estrutural que reforza a capacidade catalítica de ribozima do ARNr, que é o que realmente participa na catálise. A actividade similar aos ribozimas dos ribosomas fai pensar que son restos do "mundo de ARN", que se pensa que existiu no período da orixe da vida na Terra.[5]
Os ribosomas foron descubertos na década de 1950 polo biólogo romanés George Palade usando microscopio electrónico como partículas ou gránulos densos,[6] polo cal recibiu o premio Nobel. Pero o termo ribosoma foi proposto polo científico Richard B. Roberts en 1958, e vén de "ribo" (ribonucleico) e "soma" (corpo).
Localización
editarPoden atoparse no citosol da célula eucariótica ou procariótica ou aparecer asociados ás membranas do retículo endoplasmático rugoso ou ás da cara citoplásmica da membrana nuclear. Tamén hai ribosomas dentro das mitocondrias e cloroplastos. Tanto os ribosomas libres coma os unidos a membranas son iguais e só se diferencian na súa localización. Que un ribosoma estea libre no citosol ou unido a membranas depende da presenza dunha secuencia de sinalización que o destina ao retículo endoplasmático presente na proteína que se está empezando a sintetizar, o que quere dicir que as proteínas inician a súa síntese nos ribosomas citosólicos pero continúana no retículo endoplasmático cando levan ese sinal. Un mesmo ribosoma pode unirse ao retículo endoplasmático cando sintetiza unha proteína e non facelo se sintetiza outra proteína distinta (sen esa secuencia). Os ribosomas do retículo endoplasmático rugoso fíxanse á superficie deste orgánulo por medio dunhas proteínas da membrana do retículo endoplasmático chamadas riboforinas.
Os ribosomas libres citosólicos de 80 S poden estar en calquera parte do citosol, pero non no núcleo nin noutros orgánulos. As proteínas que se forman nestes ribosomas son liberadas no citosol e usadas dentro da célula (non son exportadas fóra). Como o citosol contén grandes cantidades de glutatión e é un ambiente redutor, as proteínas que conteñen pontes disulfuro, formadas por residuos de cisteína oxidados, non se poden sintetizar no citosol.
Nos ribosomas do retículo endoplasmático rugoso as proteínas formadas pasan ao interior das cisternas de dito retículo e son transportadas ao seu destino en vesículas pola vía secretora. Estas proteínas son proteínas de membrana destinadas xeralmente á membrana plasmática ou proteínas de exportación, que serán secretadas fóra da célula por exocitose.
Tipos de ribosomas
editarExisten varios tipos de ribosomas segundo o tipo de organismo, pero todos os ribosomas comparten a mesma estrutura central, con diferenzas de tamaño pero moi similar en todos. A maioría do ARN está moi organizado en varios motivos de estrutura terciaria, por exemplo pseudonós que mostran amoreamento coaxial. O ARN extra dos ribosomas de 80 S aparece en varias insercións continuas longas, de modo que forma bucles que saen da estrutura central sen distorsionala nin cambiala.[7] Toda a actividade catalítica do ribosoma a leva a cabo o ARNr; as proteínas están na parte superficial e parecen servir para estabilizar a estrutura.[7]
Distinguiremos os seguintes tipos de ribosomas:
Ribosomas procariotas
editarOs ribosomas das células procariotas son os máis estudados. Son de 70S, a súa masa molecular é de 2.500 kilodaltons, e o diámetro de 20 nm. As moléculas de ARNr forman o 65% do ribosoma e as proteínas representan o 35%. As moléculas de ARN ribosómico son ricas en adenina e guanina e forman unha hélice arrededor das proteínas. Os ribosomas están formados por dúas subunidades.[7] :
- Subunidade maior: é de 50S. Está formada por dúas moléculas de ARN, unha de 23S (de 2900 nucleótidos) e outra de 5S (de 120 nucleótidos). Ademais hai 34 proteínas básicas das cales só unha se repite na subunidade menor.
- Subunidade menor: é de 30S e ten unha molécula de ARNr 16S (de 1540 nucleótidos) xunto con 21 proteínas.
Os ribosomas procariotas son afectados por antibióticos que non alteran os ribosomas eucarióticos de 80S. Os ribosomas mitocondriais illados in vitro tamén se ven afectados pero na célula non, porque están protexidos pola dobre membrana do orgánulo.[8][9]
Ribosomas eucariotas
editarEn eucariotas os ribosomas son de 80S. O seu peso molecular é de 4.200 Kd e o diámetro é de 25-30 nm. Conteñen un 40% de ARNr e 60% de proteínas. Igual que os procariotas, divídense en dúas subunidades de distinto tamaño.[7]:
- Subunidade maior: é de 60 S. Ten tres tipos de ARNr: 5S (de 120 nucleótidos), 28S (de 4700 nucleótidos) e 5,8S (de 160 nucleótidos), e contén unhas 49 proteínas, todas elas distintas ás da subunidade menor.
- Subunidade menor: é de 40S. Ten unha soa molécula de ARNr de 18S (de 1900 nucleótidos) e contén unhas 33 proteínas. Dependendo da especie, este ARNr 18S pode sufrir alteracións.
Ribosomas mitocondriais
editarAs mitocondrias teñen o seu propio aparato de síntese proteica que inclúe ribosomas, ARNt e ARNm. Os ribosomas mitocondriais das células animais conteñen dous tipos de ARN ribosómico, de 12S e 16S, que se transcriben a partir de xenes do ADN mitocondrial, e son transcritos por unha ARN polimerase mitocondrial específica. Todas as proteínas que forman parte dos ribosomas mitocondriais están codificadas por xenes do núcleo celular, cuxos ARNm son traducidos no citosol e despois as proteínas transportados ata as mitocondrias.[10]
Ribosomas de plastos
editarOs ribosomas que aparecen en plastos son similares aos procariotas. Son, igual que os procariotas, de 70S, pero na subunidade maior hai un ARNr 4S que é equivalente ao de 5S procariota.
Polisomas
editarA un mesmo ARNm poden unirse varios ribosomas á vez formando en conxunto un polisoma ou polirribosoma. Deste modo o ARNm pode ser traducido por todos os ribosomas á vez, orixinándose rapidamente moitas copias da mesma proteína. Poden formarse polirribosomas cos ribosomas do retículo endoplasmático rugoso ou cos ribosomas citosólicos eucarióticos ou procarióticos.
Funcións
editarOs ribosomas son os orgánulos encargados da síntese de proteínas, proceso coñecido como tradución. A información necesaria para esa síntese encóntrase no ARN mensaxeiro (ARNm), cuxa secuencia de nucleótidos determina a secuencia de aminoácidos da proteína; pero á súa vez, a secuencia do ARNm é unha copia complementaria transcrita dun xene do ADN, que é a molécula que almacena de forma permanente a información xenética. O ARN de transferencia leva os aminoácidos aos ribosomas, onde se incorporan ao polipéptido que está sendo sintetizado.
Tradución
editar- Artigo principal: Tradución (proteínas).
O ribosoma le o ARN mensaxeiro e ensambla os aminoácidos fornecidos polos ARN de transferencia á proteína en crecemento, proceso coñecido como tradución ou síntese de proteínas.
Todas as proteínas están formadas por aminoácidos. Nas proteínas aparecen normalmente 20 aminoácidos. Os nucleótidos do ARNm, agrupados de tres en tres en todas as combinacións, forman os codóns do código xenético, cada un dos cales codifica un aminoácido. Como hai 64 codóns e só 20 aminoácidos proteicos típicos, cada aminoácido pode estar codificado por máis dun codón. Isto fai que o código sexa redundante ("dexenerado"). O código xenético non é outra cousa que as correspondencias entre cada codón e cada aminoácido. No código xenético tamén hai 3 codóns que non codifican aminoácidos e funcionan como sinais de parada da tradución.
A tradución comeza polo codón AUG no extremo 5' do ARNm, chamado codón de iniciación, que codifica o aminoácido metionina, que vai ser o primeiro aminoácido, no extremo N-terminal da proteína. As proteínas eucariotas que saen do ribosoma sempre empezan por metionina, aínda que despois poden sufrir alteracións postraducionais que a eliminan. As das bacterias empezan por N-formilmetionina. O ribosoma pode identificar o codón de iniciación grazas á secuencia Shine-Dalgarno do ARNm en procariotas ou á secuencia Kozak en eucariotas.
Despois do codón de iniciación veñen os codóns que codifican os sucesivos aminoácidos da proteína por orde, e ao final da secuencia encóntrase un codón que indica o final da tradución; é o codón de terminación, que é "mudo" (non codifica aminoácido). O código xenético é universal con poucas excepcións porque cada codón codifica o mesmo aminoácido para a gran maioría dos organismos (pode variar nun ou dous codóns en certos microorganismos e ADN mitocondrial).
Por exemplo, este ARNm: AUG-CCC-GCC-AAC-GGC-AUG-CCU-ACU-UAG
orixinaría este polipéptido: metionina-prolina-alanina-arxinina-glicina-metionina-prolina-serina,
porque esas son as correspondencias entre eses codóns cos aminoácidos e colócanse por orde. Vese como hai dous codóns (CCC e CCU) que codifican a prolina (dexeneración do código), e como o codón final UAG é un codón de parada que non codifica aminoácido.
O ribosoma presenta tres centros ou sitios de unión para os ARNt, que son os sitios A, P e E. No sitio A entra o ARNt que leva o novo aminoácido que vai ser introducido na proteína en crecemento. No sitio P está o ARNt que leva a cadea proteica en crecemento. O sitio E (de expulsión ou saída) é o lugar desde onde son expulsados do ribosoma os ARNt que xa deixaron o seu aminoácido e volven ao citosol. Cando entra un ARNt no ribosoma, que trae un novo aminoácido, toda a cadea polipeptídica formada ata ese momento se une ao novo aminoácido, formando unha cadea cun aminoácido máis. O enlace entre a cadea polipeptídica e o novo aminoácido é un enlace peptídico, que se establece grazas á actividade de peptidil transferase do ribosoma, que pode considerarse unha actividade de ribozima [11], xa que nela o ARNr xoga un papel fundamental, formando os sitios activos.
Bioxénese dos ribosomas
editarNas bacterias os ribosomas sintetízanse no citoplasma por medio da transcrición de múltiples operóns de xenes ribosómicos. Nos eucariotas a ensamblaxe das subunidades ten lugar no nucléolo, aínda que as proteínas proceden do citoplasma. O proceso de ensamblaxe das subiunidades implica o funcionamento coordinado de 200 proteínas para a síntese e procesamento dos catro ARNr necesarios, e a súa ensamblaxe entre si e coas proteínas ribosómicas.
Orixe
editarO ribosoma puido orixinarse inicialmente como un protorribosoma na Terra prebiótica,[12] que posiblemente contiña un centro de peptidil transferase (PTC), nun mundo de ARN, aparecendo como un complexo autorreplicante no que só máis tarde evolucionou a capacidade de sintetizar proteínas cando empezaron a aparecer os aminoácidos.[13] Diversos estudos suxiren que os antigos ribosomas construídos soamente con ARN ribosómico puideron ter desenvolvido a capacidade de crear enlaces peptídicos.[14][15][16][17][18] Ademais, as evidencias existentes apuntan claramente a que os antigos ribosomas eran complexos autorreplicantes, nos que o ARNr dos ribosomas tiña propósitos informacionais, estruturais e catalíticos porque posiblemente podía codificar os ARN transferentes e proteínas necesarias para a autorreplicación do ribosoma.[19] Os hipotéticos organismos celulares con ARN autorreplicante pero sen ADN denomínanse ribocitos (ou ribocélulas).[20][21]
A medida que apareceron gradualmente os aminoácidos no mundo de ARN nas condicións prebióticas,[22][23] as súas interaccións co mundo de ARN catalítico farían incrementar tanto en rango coma en eficiencia o funcionamento das moléculas de ARN catalíticas.[13] Así, a forza impulsora para a evolución do ribosoma desde unha antiga máquina autorreplicante á súa forma actual de máquina traducional puido ser a presión selectiva para incorporar proteínas aos mecanismos autorreplicantes dos ribosomas, para así incrementar a súa capacidade de autorreplicación.[19][24][25]
Notas
editar- ↑ Campbell and Reece, Biology 6th edition, Benjamin Cummings, 2002
- ↑ Cormack, David H. (1984) Introduction to Histology, Lippincott, ISBN 0-397-52114-6
- ↑ Benne R, Sloof P (1987). "Evolution of the mitochondrial protein synthetic machinery". BioSystems 21 (1): 51–68. PMID 2446672. doi:10.1016/0303-2647(87)90006-2.
- ↑ "Ribosomes". Arquivado dende o orixinal o 20 de marzo de 2009. Consultado o 4-28-2011.
- ↑ Cech T (2000). "Structural biology. The ribosome is a ribozyme". Science 289 (5481): 878–9. PMID 10960319. doi:10.1126/science.289.5481.878.
- ↑ G.E. Palade. (1955) "A small particulate component of the cytoplasm". J Biophys Biochem Cytol. Jan;1(1): pages 59-68. PMID 14381428
- ↑ 7,0 7,1 7,2 7,3 The Molecular Biology of the Cell, fourth edition. Brusce Alberts, et al. Garland Science (2002) pg. 342 ISBN 0-8153-3218-1
- ↑ Recht MI, Douthwaite S, Puglisi JD (1999). "Basis for bacterial specificity of action of aminoglycoside antibiotics". EMBO J 18 (11): 3133–8. PMC 1171394. PMID 10357824. doi:10.1093/emboj/18.11.3133.
- ↑ O'Brien, T.W., The General Occurrence of 55S Ribosomes in Mammalian Liver Mitochondria. J. Biol. Chem., 245:3409 (1971)
- ↑ Devlin, T. M. 2004. Bioquímica, 4ª edición. Reverté, Barcelona. ISBN 84-291-7208-4
- ↑ Rodnina MV, Beringer M, Wintermeyer W (2007). "How ribosomes make peptide bonds". Trends Biochem. Sci. 32 (1): 20–6. PMID 17157507. doi:10.1016/j.tibs.2006.11.007.
- ↑ Dance, Amber (28 de febreiro 2023). "How did life begin? One key ingredient is coming into view - A Nobel-prizewinning scientist's team has taken a big step forward in its quest to reconstruct an early-Earth RNA capable of building proteins.". Nature 615 (7950): 22–25. PMID 36854922. doi:10.1038/d41586-023-00574-4.
- ↑ 13,0 13,1 Noller HF (abril de 2012). "Evolution of protein synthesis from an RNA world". Cold Spring Harbor Perspectives in Biology 4 (4): a003681. PMC 3312679. PMID 20610545. doi:10.1101/cshperspect.a003681.
- ↑ Dabbs ER (1986). Mutant studies on the prokaryotic ribosome. Nova York: Springer-Verlag.
- ↑ Noller HF, Hoffarth V, Zimniak L (xuño de 1992). "Unusual resistance of peptidyl transferase to protein extraction procedures". Science 256 (5062): 1416–9. Bibcode:1992Sci...256.1416N. PMID 1604315. doi:10.1126/science.1604315.
- ↑ Nomura M, Mizushima S, Ozaki M, Traub P, Lowry CV (1969). "Structure and function of ribosomes and their molecular components". Cold Spring Harbor Symposia on Quantitative Biology 34: 49–61. PMID 4909519. doi:10.1101/sqb.1969.034.01.009.
- ↑ Krupkin M, Matzov D, Tang H, Metz M, Kalaora R, Belousoff MJ, Zimmerman E, Bashan A, Yonath A (Oct 2011). "A vestige of a prebiotic bonding machine is functioning within the contemporary ribosome". Phil. Trans. R. Soc. B 366 (1580): 2972–8. PMC 3158926. PMID 21930590. doi:10.1098/rstb.2011.0146.
- ↑ Bose T, Fridkin G, Davidovich C, Krupkin M, Dinger N, Falkovich AH, Peleg Y, Agmon I, Bashan A, Yonat A (febreiro de 2022). "Origin of life: protoribosome forms peptide bonds and links RNA and protein dominated worlds". Nucleic Acids Res 50 (4): 1815–1828. PMC 8886871. PMID 35137169. doi:10.1093/nar/gkac052.
- ↑ 19,0 19,1 Root-Bernstein M, Root-Bernstein R (febreiro de 2015). "The ribosome as a missing link in the evolution of life". Journal of Theoretical Biology 367: 130–158. PMID 25500179. doi:10.1016/j.jtbi.2014.11.025.
- ↑ Yarus M (2002). "Primordial genetics: phenotype of the ribocyte". Annual Review of Genetics 36: 125–51. PMID 12429689. doi:10.1146/annurev.genet.36.031902.105056.
- ↑ Forterre P, Krupovic M (2012). "The Origin of Virions and Virocells: The Escape Hypothesis Revisited". Viruses: Essential Agents of Life. pp. 43–60. ISBN 978-94-007-4898-9. doi:10.1007/978-94-007-4899-6_3.
- ↑ Caetano-Anollés G, Seufferheld MJ (2013). "The coevolutionary roots of biochemistry and cellular organization challenge the RNA world paradigm". Journal of Molecular Microbiology and Biotechnology 23 (1–2): 152–77. PMID 23615203. doi:10.1159/000346551.
- ↑ Saladino R, Botta G, Pino S, Costanzo G, Di Mauro E (agosto de 2012). "Genetics first or metabolism first? The formamide clue". Chemical Society Reviews 41 (16): 5526–65. PMID 22684046. doi:10.1039/c2cs35066a. hdl:11573/494138.
- ↑ Fox GE (setembro de 2010). "Origin and Evolution of the Ribosome". Cold Spring Harb Perspect Biol 2 (9): a003483. PMC 2926754. PMID 20534711. doi:10.1101/cshperspect.a003483.
- ↑ Fox GE (2016). "Origins and early evolution of the ribosome". En Hernández G, Jagus R. Evolution of the Protein Synthesis Machinery and Its Regulation. Suíza: Springer, Cham. pp. 31–60. ISBN 978-3-319-39468-8. doi:10.1007/978-3-319-39468-8.
Véxase tamén
editarOutros artigos
editar- Bóveda (orgánulo), outro orgánulo ribonucleoproteico.
- Péptido non ribosómico, péptidos producidos encimaticamente sen intervención dos ribosomas.
Ligazóns externas
editar- 70S Ribosome Architecture Animation of a working ribosome. Requires the Chime browser plugin from this site (where registration is required).
- Lab computer simulates ribosome in motion
- Role of the Ribosome, Gwen V. Childs, copied here
- Ribosome in Proteopedia - The free, collaborative 3D encyclopedia of proteins & other molecules
- Ribosomal proteins families in ExPASyArquivado 30 de abril de 2011 en Wayback Machine.
- Molecule of the MonthArquivado 27 de outubro de 2009 en Wayback Machine. © RCSB Protein Data Bank:
- RibosomeArquivado 14 de novembro de 2010 en Wayback Machine.
- Elongation FactorsArquivado 16 de marzo de 2011 en Wayback Machine.
- Palade