Estructura de les proteínes
La estructura de les proteínes axunta les propiedaes de disposición nel espaciu de les molécules de proteína que provienen de la so secuencia d'aminoácidos, les carauterístiques físiques de la so redolada y la presencia de compuestos simples o complexos que les estabilicen y conduzan a un plegamiento específicu.
Estructura de los aminoácidos
editarLos aminoácidos, monómeros componentes del polímeru proteína, son molécules quirales constituyíes por un átomu de carbonu central, el Cα, que porten nésti un grupu amino y un grupu carboxilo, lo cual da-yos el so nome, amás d'un átomu d'hidróxenu y una cadena llateral que-yos confier les sos carauterístiques definitories, y en función de la cual clasifíquense.[1]
Los 20 α-L-aminoácidos proteinogénicos son los llistaos na siguiente tabla:
| Nome | Códigu de trés lletres |
Códigu d'una lletra |
Bayura relativa (%) E.C. |
MW | pK | VdW volume (ų) |
Cargado, Polar, Hidrofóbico, Neutro |
|---|---|---|---|---|---|---|---|
| Alanina | Ala | A | 13.0 | 71 | 67 | H | |
| Arginina | Arg | R | 5.3 | 157 | 12.5 | 148 | C+ |
| Asparagina | Asn | N | 9.9 | 114 | 96 | P | |
| Aspartato | Asp | D | 9.9 | 114 | 3.9 | 91 | C- |
| Cisteína | Cys | C | 1.8 | 103 | 86 | P | |
| Glutamato | Glu | E | 10.8 | 128 | 4.3 | 109 | C- |
| Glutamina | Gln | Q | 10.8 | 128 | 114 | P | |
| Glicina | Gly | G | 7.8 | 57 | 48 | N | |
| Histidina | His | H | 0.7 | 137 | 6.0 | 118 | P, C+ |
| Isoleucina | Ile | I | 4.4 | 113 | 124 | H | |
| Leucina | Leu | L | 7.8 | 113 | 124 | H | |
| Lisina | Lys | K | 7.0 | 129 | 10.5 | 135 | C+ |
| Metionina | Met | M | 3.8 | 131 | 124 | H | |
| Fenilalanina | Phe | F | 3.3 | 147 | 135 | H | |
| Prolina | Pro | P | 4.6 | 97 | 90 | H | |
| Serina | Ser | S | 6.0 | 87 | 73 | P | |
| Treonina | Thr | T | 4.6 | 101 | 93 | P | |
| Triptófanu | Trp | W | 1.0 | 186 | 163 | P | |
| Tirosina | Tyr | Y | 2.2 | 163 | 10.1 | 141 | P |
| Valina | Val | V | 6.0 | 99 | 105 | H |
Termodinámica del plegamientu
editarEn condiciones fisiolóxiques, el procesu de plegamientu d'una proteína globular ta claramente favorecíu termodinámicamente, esto ye, la medría d'enerxía llibre global del procesu tien de ser negativu (anque dalgún de los sos pasos puede ser positivu). Esti cambéu consíguese permediando una serie de factores termodinámicos como la entropía conformacional, les interacciones carga-carga, los puente d'hidróxenu internos, les interacciones hidrofóbiques y les interacciones de van der Waals.[2] .[3]
Entropía conformacional
editarDefínese entropía conformacional del plegáu como l'amenorgamientu de la entropía, de l'aleatoriedad a última hora, mientres el pasu dende un ensame de conformances de duviellu aleatoriu hasta una única estructura plegada. La enerxía llibre, representada na ecuación ΔG = ΔH - T ΔS, demuestra que'l ΔS negativu realiza una contribución positiva a ΔG. Esto ye, el cambéu d'entropía conformacional oponer al plegáu. Por ello, el ΔG global, que tien de ser negativu, deber a qu'o bien ΔH ye negativu y grande o a dalgún otru aumentu de la entropía col plegáu. Na práutica dan dambes coses.[3]
La fonte de ΔH negativu ye'l cúmulu d'interacciones favorables energéticamente que se dan nel interior del glóbulu proteicu, interacciones que suelen ser non covalentes.
Interacciones carga-carga
editarLes interacciones carga-carga dar ente grupos polares y cargaos de les cadenes llaterales de los aminoácidos componentes del polipéptido, yá que los grupos carboxilo y amino del carbonu alfa tán implicaos nel enllaz peptídico. D'esta miente, dichos grupos ionizados atráense y formen un equivalente a sales ente residuos del polipéptido: ello ye que denominar dacuando pontes salines.[3] Evidentemente, diches interacciones sumen cuando'l pH del mediu ye tal que se pierde l'estáu d'ionización del grupu; ello ye que esta ye una de les causes de la desnaturalización rápida de les proteínes por aciu adición d'ácidos o bases, y apodera a les proteínes a una redolada d'un pH tamponado y moderáu, que ye'l fisiolóxicu, salvo esceiciones, como pue ser l'interior lisosomal na redolada subcelular[4]
Enllaces d'hidróxenu internos
editarLes cadenes llaterales de munchos aminoácidos pórtense como apurridores o como aceptores d'enllaces d'hidróxenu (ye'l casu de los grupos hidroxilo de la serina y los grupos amino de la glutamina, por casu). Amás, si los protones amida o los carbonilos del armazón polipeptídico nun tán implicaos nel enllaz peptídico, pueden interaccionar tamién nesti tipu d'uniones estabilizadores.
Magar los enllaces d'hidróxenu son débiles en disolución aguacienta.[3] el so gran númberu puede estabilizar, y facer, la estructura terciaria proteica.
Interacciones de van der Waals
editarEl trupu empaquetamiento nel nucleu de les proteínes globulares facilita la interacción débil ente grupos moleculares ensin carga. Dichos enllaces son de baxa enerxía, pero'l so abondosu númberu suple la so debilidá. Cada interacción individual namái contribúi nunos pocos quiloxulios a la entalpía d'interacción negativa global. Pero la suma de toles contribuciones de toles interacciones sí que puede estabilizar a la estructura plegada. D'esta miente, una contribución enerxética favorable a partir de la suma de les interacciones intramoleculares compensa de manera más qu'abonda la entropía desfavorable del plegáu.[3]
Interacciones hidrofóbiques
editarPor definición, cualquier sustancia hidrofóbico en contautu col agua provoca qu'ésta fuxa y arrexuntar n'estructures denominaes clatratos.[4] Esta ordenación correspuende a un amenorgamientu de la entropía del sistema. Nel casu de les proteínes, los residuos hidrofóbiques de los aminoácidos queden empobinaos, nel so plegamientu, escontra l'interior de la molécula, en contautu colos sos asemeyaos y alloñaos de l'agua. Arriendes d'ello, la internalización de los grupos hidrófobos aumenta la aleatoriedad del sistema 'proteína más agua' y, poro, produz un aumentu d'entropía al doblar. Esti aumentu d'entropía produz una contribución negativa a la enerxía llibre del plegáu y aumenta la estabilidá de la estructura proteica.[3]
Función de los enllaces disulfuro
editarLa estabilización de la proteína recién plegada puede asoceder por aciu la formación de pontes disulfuro ente residuos de cisteína axacentes o engarraos. Dichu procesamientu producir en munchos casos nel lumen del retículo endoplasmático por aciu la enzima disulfuro isomerasa, presente en toles célules eucariotes. Dicha enzima, que cataliza la oxidación de los grupos sulfhidrilo o grupos tiol (-SH2) de los residuos de cisteína, ye especialmente abondosa n'órganos como'l fégadu o páncrees, onde se producen pequeñes cantidaes de proteínes que contienen esti tipu d'enllaces.[5]
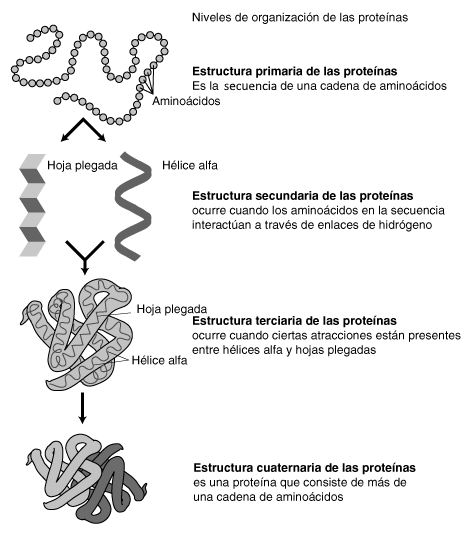
Niveles de estructuración
editarLa estructura de les proteínes puede xerarquizase nuna serie de niveles, interdependientes. Estos niveles correspuenden a:
- Estructura primaria, que correspuende a la secuencia d'aminoácidos.
- Estructura secundaria, que provoca l'apaición de motivos estructurales.
- Estructura terciaria, que define la estructura de les proteínes compuestes por un namái polipéptido.
- Estructura cuaternaria, si intervién más d'un polipéptido.
Estructura primaria
editarLa estructura primaria de les proteínes referir a la secuencia d'aminoácidos, esto ye, la combinación llinial de los aminoácidos por aciu un tipu d'enllaz covalente, l'enllaz peptídico. Los aminoácidos tán xuníos por enllaces peptídicos siendo una de les sos carauterístiques más importantes la coplanaridad de los radicales constituyentes del enllaz.
La estructura llinial del péptidu va definir en gran midida les propiedaes de niveles d'organización cimeres de la proteína. Esti orde ye consecuencia de la información del material xenético: Cuando se produz la traducción del RNA llógrase l'orde d'aminoácidos que van dar llugar a la proteína. Puede dicise, por tanto, que la estructura primaria de les proteínes nun ye más que l'orde d'aminoácidos que lo conformen.
Estructura secundaria
editarLa estructura secundaria de les proteínes ye la disposición espacial llocal de la cadarma proteica, gracies a la formación de pontes d'hidróxenu ente los átomos que formen l'enllaz peptídico, esto ye, un tipu d'enllaz non covalente, ensin faer referencia a la cadena llateral. Esisten distintos tipos d'estructura secundaria: - Estructura secundaria ordenada, ( repetitivos onde s'atopen los hélices alfa y cadenes beta, y non repetitivos onde s'atopen los xiros beta y comba beta) -Estructura secundaria non ordenar -Estructura secundaria desordenada
Los motivos más comunes son la héliz alfa y la beta llámina (Fueya plegada beta).
- Héliz alfa Los
aminoácidos nuna héliz α tán dispuestos nuna estructura helicoidal dextrógira, con unos 3.6 aminoácidos per vuelta. Cada aminoácidu supón un xiru d'unos 100° na héliz, y los carbonos α de dos aminoácidos allegantes tán dixebraos por 1.5Å. La héliz ta estrechamente empaquetada, de forma que nun hai cuasi espaciu llibre dientro de la héliz. Toles cadenes llaterales de los aminoácidos tán dispuestes escontra l'esterior de la héliz.[6]
El grupu amino del aminoácidu (n) puede establecer un enllaz d'hidróxenu col grupu carbonilo del aminoácidu (n+4). D'esta forma, cada aminoácidu (n) de la héliz forma dos pontes d'hidróxenu col so enllaz peptídico y l'enllaz peptídico del aminoácidu en (n+4) y en (n-4). En total son 7 enllaces d'hidróxenu per vuelta. Esto estabiliza descomanadamente la héliz. Ta dientro de los niveles d'organización de la proteína.
- Llámina beta La beta llámina formar
pol allugamientu paralelu de dos cadenes d'aminoácidos dientro de la mesma proteína, nel que los grupos amino d'una de les cadenes formen enllaces d'hidróxenu colos grupos carboxilo de la opuesta. Ye una estructura bien estable que puede llegar a resultar d'una rotura de los enllaces d'hidróxenu mientres la formación de la héliz alfa. Les cadenes llaterales d'esta estructura tán asitiaos sobre y sol planu de les llámines. Dichos sustituyentes nun tienen de ser bien grandes, nin crear un torga estérico, yá que se vería afeutada la estructura de la llámina.[7]
Estructura terciaria
editarYe la manera en que la cadena polipeptídica plegar nel espaciu, esto ye, cómo s'endolca una determinada proteína, yá seya globular o fibrosa. Ye la disposición de los dominios nel espaciu.
La estructura terciaria realízase de manera que los aminoácidos apolares asitiar escontra l'interior y los polares escontra l'esterior en medios aguacientos. Esto provoca una estabilización por interacciones hidrofóbiques, de fuercies de van der Waals y de pontes disulfuro[1] (covalentes, ente aminoácidos de cisteína convenientemente empobinaos) y por aciu enllaces iónicos.
Estructura cuaternaria
editarLa estructura cuaternaria deriva de la conxunción de delles cadenes peptídicas que, acomuñaes, conformen un ente, un multímero, que tien propiedaes distintes a la de les sos monómeros componentes. Estes subunidades acomuñar ente sigo por aciu interacciones non covalentes, como pueden ser pontes d'hidróxenu, interacciones hidrofóbiques o pontes salines. Pal casu d'una proteína constituyida por dos monómeros, un dímero, ésti pue ser un homodímero, si los monómeros constituyentes son iguales, o un heterodímero, si nun lu son.
Ángulos de rotación y representaciones de Ramachandran
editarNuna cadena polipeptídica, defínense dos enllaces del armazón capaces de rotar: unu ye l'enllaz ente'l nitróxenu y el Cα, y l'otru l'enllaz ente'l Cα y l'osíxenu del carbonilo. Dambos definen dos ángulo de rotación:
- L'ángulu de rotación φ, definíu polos cuatro átomos socesivos de la cadarma CO-NH-Cα-CO, implicando a dos aminoácidos.
- L'ángulu de rotación ψ, definíu polos cuatro átomos socesivos de la cadarma: NH-Cα-CO-NH, qu'implica a dos aminoácidos.
La orientación de la exa de xiru considerada positiva por convención amosar na imaxe; correspuende a la mesma de les manes del reló.[3]
Esisten dos esceición nos aminoácidos que se representen nestes diagrames: la glicina, carente d'un sustituyente, y la prolina, cíclica por cuenta de la tenencia d'una estructura tipu pirrol, nun cumplen los requisitos riquíos pa una representación convencional[8]
Puede describise, poro, la conformanza del armazón de cualquier residuu concreta d'una proteína especificando estos dos ángulos.[3] Con estos dos parámetros podemos describir la conformanza de dichu residuu nun mapa por aciu un puntu, con coordenaes φ y ψ. Pa determinaos tipos d'estructura secundaria, como la héliz alfa, toles residuos comparten dichos ángulos, polo qu'un puntu nel mapa en determinada posición puede describir una estructura secundaria. Estos mapes, denominaos representaciones de Ramachandran, pol bioquímicu G. N. Ramachandran[9] que los usó por llargamente en [1963], dexen deducir diches conformances.[3]
Dominios, motivos y otros elementos conformacionales
editarYe común que delles zones de la proteína tengan entidá estructural independiente, y de cutiu funciones bioquímiques específiques, como, por casu, dalguna actividá catalítica. La so naturaleza depende de les estructures enantes citaes a tolos niveles.
La estructura cuaternaria deriva de la conxunción de delles cadenes peptídicas que, acomuñaes, conformen un ente, un multímero, con propiedaes distintes.
Les proteínes tán entamaes en munches unidaes. Un dominiu estructural ye un elementu de la estructura de les proteínes que se autoestabiliza y de cutiu estabiliza a los motivos conformacionales independientemente del restu de la cadena de proteína. Munchos dominios son únicos y vienen de una secuencia única d'un xen o una familia xénica pero sicasí otros apaecen nuna variedá de proteínes. Los dominios son, de cutiu, escoyíos evolutivamente porque tienen una función prominente na bioloxía de la proteína pertenecen; por casu, "l'apodero d'unión a calciu de calmodulina". La inxeniería xenética dexa modificar los dominios d'una proteína a otra pa xenerar proteínes quimériques con funciones novedoses.[10] Un motivu nesti sentíu referir a una combinación específica d'elementos estructurales secundarios (como los héliz-xiru-héliz). Estos elementos son llamaos de cutiu superestructuras secundaries.
Suel denominase motivu conformacional de forma global a un tipu de motivu, como los barriles-beta. La estructura de los motivos de cutiu consiste en solo unos pocos elementos, por casu, les héliz-xiru-héliz, que namái tienen trés. Se denota que la secuencia espacial” ye la mesma en toles instancies del motivu. El so orde ye abondo irregular dientro del xen subxacente. Los motivos estructurales de la proteína de cutiu inclúin xiros de llargor variable n'estructures indeterminaes, lo que n'efeutu crea la plasticidad necesaria pa xunir dos elementos nel espaciu que nun tán codificados por una secuencia d'ADN darréu axacente nun xen. Se denota tamién qu'inclusive cuando tán codificados los elementos estructurales secundarios d'un motivu nel mesmu orde en dos xenes, la composición cuantitativa d'aminoácidos puede variar. Esto non yá ye ciertu por cuenta de les complicaes rellaciones ente la estructura terciario y primario, sinón por cuestiones relatives al tamañu. Magar na base de datos de lleldu hai descrites unes 6.000 proteínes,[11] hai munchos menos dominios, motives estructurales y plegues. Esto ye, en parte, consecuencia de la evolución. Esto significa, por casu, qu'un dominiu d'una proteína puede ser treslladáu d'una a otra, dando asina una nueva función a les proteínes. Por cuenta de estos mecanismos, los dominios o motivos estructurales pueden ser comunes a delles families de proteínes.
Cinética del plegáu de les proteínes
editarAnque'l plegamientu de les proteínes parta d'un estáu llinial inicial y unu final bien definíos, el procesu de plegamientu nun ye daqué sópitu con un llugar de partida y un fin, sinón que ta llaráu d'entemedios temporalmente mensurables y de vital importancia. Inclusive, la estructura final falta enforma de ser estática: dellos autores imaxinen a les proteínes como entidaes dinámiques que de cutio camuden d'estructura, d'una manera similar al llatíu cardiacu[12]
Magar el plegamientu d'una proteína ye un sucesu rápidu, que se completa n'apenes un segundu, topológicamente ye un problema bien complexu. Esti fechu dio llugar a la paradoxa de Levinthal, mesma de Cyrus Levinthal en 1968: un cálculu averáu indica qu'una cadena polipeptídica d'unos 120 residuos tien unes 1050 conformances. Anque la molécula pudiera intentar una nueva conformanza cada 10-13 segundos, precisaríense unos 1030 años pa intentar un númberu significativu d'elles.[3] Sicasí, proteínes d'estes carauterístiques pliéguense in vitro nun minutu.
La resolución de la paradoxa pasa pola aceptación de la esistencia d'estaos de plegamientu entemedios, nos que la proteína atópase parcialmente esplegada, nuna ruta como sigue:[3]
- Proteína esplegada.
- Nucleación del plegáu.
- Estaos entemedios.
- Estáu de glóbulu fundíu.
- Reordenamientos finales.
- Proteína plegada.
Elementos moduladores
editarTemperatura
editarEl papel de la temperatura ye crucial yá que la so entidá físicu-química, la enerxía cinética contenida nos átomos, dota de reactividá a los aminoácidos y, por ello, a les proteínes. Sicasí, esiste una llende, a unos 50 °C, devasáu'l cual les proteínes pierden la so conformanza, esto ye, se desnaturalizan.
La desnaturalización, producida pola temperatura y otros axentes desnaturalizantes, asocede a dellos niveles:
- Na desnaturalización de la estructura cuaternaria, les subunidades de proteínes dixébrense o la so posición espacial malvar.
- La desnaturalización de la estructura terciaria implica la interrupción de:
- Enllaces covalentes ente les cadenes llaterales de los aminoácidos (como les pontes disulfuro ente les cisteínas).
- Enllaces non covalentes dipolo-dipolo ente cadenes llaterales polares d'aminoácidos.
- Enllaces dipolo inducíos por fuercies de Van Der Waals ente cadenes llaterales non polares d'aminoácidos.
- Na desnaturalización de la estructura secundaria les proteínes pierden tolos patrones de repetición regulares como les alfa hélizs y adopten formes aleatories.
- La estructura primaria, la secuencia d'aminoácidos amestaos por enllaces peptídicos, nun ye atayada poles desnaturalización.
pH
editarEl pH afecta al estáu iónicu de los aminoácidos, zwitteriones a última hora, que nun tienen implicáu'l so grupu amino nin carboxilo nel enllaz peptídico y, especialmente, a aquéllos polares, con dalgún grupu cargáu na so cadena llateral. L'estáu d'ionización d'ésti afecta a la reactividá y posibilidá, por tanto, de producir un enllaz químicu.[1]
Chaperonas
editarLes proteínes tipu chaperona son un conxuntu de proteínes presentes en toles célules que la so función ye la d'ayudar al plegamientu d'otres proteínes, tres la so síntesis o mientres el so ciclu d'actividá (por casu, en defensa de estrés térmicu).[4] Estes chaperonas nun formen parte de la estructura primaria de la proteína funcional, sinón que namái se xunen a ella p'ayudar nel so plegamientu, ensamblaxe y tresporte celular a otra parte de la célula onde la proteína realiza la so función. Los cambeos de conformanza tridimensional de les proteínes pueden tar afeutaos por un conxuntu de delles chaperonas que trabayen coordinaes, dependiendo de la so propia estructura y de la disponibilidad de les chaperonas.[13]
Esisten sustancies químiques non proteiques que pueden estabilizar a les proteínes por aciu l'establecimientu d'enllaces de ponte d'hidróxenu, en sustitución a los de l'agua. Por casu, ye'l casu de la trehalosa, un azucre que s'emplega en bioteunoloxía pa favorecer la viabilidá de les soluciones riques en proteína inclusive en condiciones de estrés ambiental[14]
Les chaperonas, alcontraes en tolos compartimientos celulares, arrexuntar en dos families xenerales.[4]
- Chaperonas moleculares
Que se xunen y estabilicen a proteínes esplegaes o parcialmente plegaes, evitando asina que s'arrexunten y que sían degradaes. Integren la familia de les Hsp70 nel citosol y la matriz de la mitocondria, BiP nel retículo endoplasmático y DnaK nes bacteries.
- Chaperoninas
Que faciliten directamente'l plegáu de les proteínes. Inclúin a: TriC, n'eucariotes; y GroEL, en bacteries y cloroplastos,
Clasificación estructural
editarMunchos métodos fueron desenvueltos pa la clasificación estructural de les proteínes; la escoyeta de datos ye almacenada nel Bancu de Datos de Proteínes. Munches bases de datos esistentes clasifiquen les proteínes usando distintos métodos. El SCOP, CATH y FSSP son los más usaos.
Los métodos usaos podríen clasificase en: puramente manuales, manuales y automáticos y puramente automáticos. El mayor problema d'estos métodos ye la integración de los datos. La clasificación ye constante ente SCOP, CATH Y FSSP pa la mayoría de les proteínes que fueron clasificaes, pero hai inda delles diferencies ya inconsistencies.
Determinación de la estructura proteica
editarAlredor del 90% de les estructures de les proteínes disponibles nel Bancu de Datos de Proteínes fueron determinaes por cristalografía de rayos X. Esti métodu dexa midir la densidá de distribución de los electrones de la proteína nes 3 dimensiones (nel estáu de cristalización), lo que dexa llograr les coordenaes 3D de tolos átomos pa determinar la so posición con certidume. Aproximao'l 9% de les estructures de proteínes conocíes fueron llograes por téuniques de resonancia magnética nuclear, que tamién pueden ser usaes pa identificar estructures secundaries.
Nótese que los aspeutos de les estructures secundaries pueden ser detectaos por aciu medios bioquímicos como'l dicroísmo circular[15] El microscopiu crioelectrónico convirtióse apocayá nun mediu pa determinar les estructures proteiques con una resolución baxu (menos de 5 Å ó 0,5 nm) y prevese que va ser una ferramienta importante pa los trabayos d'alta resolución na década próxima. Esta téunica ye entá un recursu importante pa científicos que tán trabayando en complexos bien grandes de proteínes, como la cubierta y cápside de los virus y les proteínes amiloideas.[16][17]
| Resolvimientu | Interpretación |
| >4.0 | Coordenaes individuales ensin significáu |
| 3.0 - 4.0 | Plegamientu posiblemente correutu, pero comúnmente con errores. Delles cadenes llaterales tienen mal los rotámeros. |
| 2.5 - 3.0 | Plegamientu bien resueltu salvu en delles plegues superficiales, mal modelaos. Delles cadenes llaterales llargues (Lys, Glu, Gln) y otres curties (Ser, Val, Thr) mal empobinaes. |
| 2.0 - 2.5 | El númberu de cadenes llaterales con un rotámero incorreutu ye enforma menor. Los errores, pequeños, son detectaos de normal. Les plegues superficiales tán abondo bien definíos. Los ligandos y l'agua son visibles. |
| 1.5 - 2.0 | Poques residuos tienen mal rotámero. Los errores pequeños son detectaos. Les plegues incorreutes son bien raros, inclusive en superficie. |
| 0.5 - 1.5 | Polo xeneral, tou ta correutamente resueltu. Les llibreríes de rotámeros y los estudios xeométricos facer a esti nivel de precisión. |
Investigación
editarEsisten ensame de grupos qu'investiguen tou lo rellacionao cola determinación de la estructura proteica, la so termodinámica, cinética ya implicaciones. Estes postreres son d'especial relevancia en neuropatoloxía, yá que enfermedaes dexeneratives como'l Alzheimer[18] o infeicioses como'l Creutzfeldt-Jacob[19] tienen la so causa n'alteraciones na estructura de les proteínes.
Les ferramientes d'investigación son, en munchos casos, simulaciones de plegamientu por aciu programes informáticos. Los proyeutos de mayor actividá tocantes a computación distribuyida son:
- Docking@home (Universidá de Texas)
- Folding@home (Universidá de Stanford)
- Proteins@home (Escuela Politéunica de Francia)
- Rosetta@home (Universidá de Washington)
Ver tamién
editarReferencies
editar- ↑ 1,0 1,1 1,2 Lehninger, Albert (1993). Principles of Biochemistry, 2nd Ed.. Worth Publishers. ISBN 0-87901-711-2.
- ↑ «Empirical scale of side-chain conformational entropy in protein folding», J Mol Biol 231 (3), doi:, http://www.ncbi.nlm.nih.gov/pubmed/8515453
- ↑ 3,00 3,01 3,02 3,03 3,04 3,05 3,06 3,07 3,08 3,09 3,10 Mathews, C. K.; Van Holde, K.Y et Ahern, K.G (2003). «6», Bioquímica, 3ª, páx. 204 y ss. ISBN 84-7892-053-2.
- ↑ 4,0 4,1 4,2 4,3 Lodish et al. (2005). Biología celular y molecular. Buenos Aires: Médica Panamericana. ISBN 950-06-1974-3.
- ↑ Sela M, Lifson S. (1959). «The reformation of disulfide bridges in proteins». Biochim Biophys Acta 36 (2): páxs. 471-8. doi:. PMID 14444674.
- ↑ Neurath, H (1940). «Intramolecular folding of polypeptide chains in relation to protein structure». Journal of Physical Chemistry 44: páxs. 296-305. doi:.
- ↑ Voet, Donald; Voet, Judith G. (2004). Biochemistry, 3rd, Hoboken, NJ: Wiley, páx. 227-231. ISBN 047119350X.
- ↑ Carl-Ivar Brändén, John Tooze (trad. Bernard Lubochinsky, préf. Joël Janin), Introduction à la structure des protéines, De Boeck Université, Bruxelles, 1996 ISBN 2-8041-2109-7
- ↑ Ramachandran, G.N., Sasisekharan, V. & Ramakrishnan, C. (1963) J. Mol. Biol. 7, 95–99 (1963
- ↑ Casado-Vela, J. (12 d'avientu de 2012). «Protein chimerism: Novel source of protein diversity in humans adds complexity to bottom-up proteomics». Proteomics 13 (1): páxs. 5-11. doi:. http://onlinelibrary.wiley.com/doi/10.1002/pmic.201200371/abstract;jsessionid=3F182057ABC8C87C5F204357AE932AEE.d01t04.
- ↑ «Yeast Protein database (YPD): a database for the complete proteome of Saccharomyces cerevisiae», Nucleic Acids Research 25 (1), doi:, PMID 9016505, http://nar.oxfordjournals.org/cgi/content/abstract/25/1/57
- ↑ Alberts et al (2004). Biología molecular de la célula. Barcelona: Omega. ISBN 54-282-1351-8.
- ↑ Ellis RJ, van der Vies SM (1991). «Molecular chaperones». Annu. Rev. Biochem. 60: páxs. 321-47. doi:. PMID 1679318.
- ↑ Prescott, L.M. (199). Microbiología. McGraw-Hill Interamericana d'España, S.A.U.. ISBN 84-486-0261-7.
- ↑ Juan Antonio Llugo-Ríos. «Dicroismo circular». Consultáu'l 8 d'ochobre de 2007.
- ↑ «[http://www.nature.com/nature/journal/v371/n6494/abs/371261a0.html Location of a folding protein and shape changes in GroEL�GroES complexes imaged by cryo-electron microscopy]», Nature 371 (6494), doi:, http://www.nature.com/nature/journal/v371/n6494/abs/371261a0.html
- ↑ «Cryo-electron microscopy of viruses», Nature 308 (5954), doi:, http://www.nature.com/nature/journal/v308/n5954/abs/308032a0.html
- ↑ Hashimoto M, Rockenstein Y, Crews L, Masliah Y (2003). «Role of protein aggregation in mitochondrial dysfunction and neurodegeneration in Alzheimer's and Parkinson's diseases.». Neuromolecular Med 4 (1-2): páxs. 21-36. PMID 14528050.
- ↑ Baker & Ridley (1996). Prion Disease. New Jersey: Humana Press. 0-89603-342-2.