Recombinação homóloga
A recombinação genética homóloga ocorre entre sequências precisamente correspondentes, de modo que nenhum par de bases é adicionado ou perdido. A recombinação que envolve uma reação entre sequências homólogas de DNA é denominada Recombinação Homóloga. Além dessa, existem pelo menos outras duas recombinações genéticas: a Recombinação Sítio-Específica e a Transposição[1].

A recombinação homóloga acontece em eucariotos, durante a prófase I da meiose que forma os gametas maternos e paternos. Na meiose I, as duas cópias de cada cromossomo podem ter diferentes alelos em algum lócus. Por meio da recombinação homóloga cromossomos recombinantes são gerados e são diferentes dos cromossomos parentais. O resultado da recombinação é a troca física de material dos cromossomos, esse evento também é conhecido como crossing-over[1]. Uma simplificação desse evento pode ser vista na figura 1.
A meiose começa com uma célula que tem seus cromossomos duplicados, de forma que existem quatro cópias de cada cromossomo no estágio inicial. O início dessa divisão é caracterizado por tornar os cromossomos mais visíveis. Cada um desses cromossomos foi replicado e consiste em duas cromátides irmãs, cada uma contendo um DNA duplex. Os cromossomos homólogos se aproximam e começam a parear, formando bivalentes. O pareamento se estende até que o comprimento total de cada cromossomo esteja alinhado a seu homólogo. Esse processo é denominado sinapse ou pareamento cromossomal. Quando o processo é finalizado, os cromossomos se associam lateralmente formando o complexo sinaptonêmico[1].
A complementariedade das duas fitas de DNA é essencial no processo de recombinação. Cada uma das cromátides irmãs consiste em um duplex de DNA, para que eles sejam quebrados e reconectados sem qualquer perda de material genético, é necessário um mecanismo capaz de reconhecer exatamente as posições correspondentes. Isso é fornecido pelo pareamento de bases complementares.


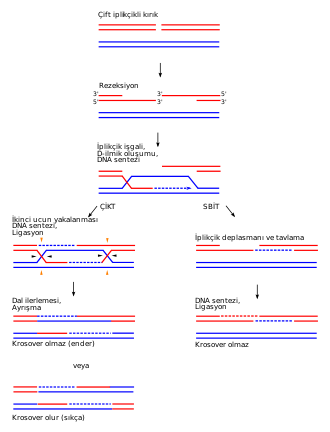
A recombinação homóloga é iniciada por uma endonuclease que cliva um dos DNAs duplexes participantes, o “receptor”. O ponto de clivagem é aumentado, gerando uma lacuna. As exonucleases digerem uma fita em um dos lados da clivagem, gerando terminações 3’ de fita simples. Então uma das extremidades 3’ livre invade uma região homóloga do outro DNA duplex em outro cromossomo, o “doador”. A formação de um DNA heteroduplex gera a alça D, na qual uma fita do doador é deslocada. A alça D é estendida pela síntese de DNA, empregando a extremidade 3’ livre como um iniciador para gerar DNA dupla fita. Eventualmente, a alça D se torna grande o suficiente para corresponder à extensão completa da lacuna no DNA duplex “receptor”[1]. Essa síntese desloca uma fita do duplex “doador” que migra para o outro duplex e serve como molde no duplex “receptor”, resultando na síntese de uma nova sequência a partir da extremidade 3’ da fita receptora usando a fita doadora como molde. As sínteses ocorrem sempre a partir da extremidade 3’. Ocorre a migração recíproca da extremidade oposta à da fita receptora, gerando um duplo crossover. Quando a fita simples projetada no DNA “doador” alcança a outra ponta da lacuna, as sequencias complementares das fitas simples se anelam e formam um DNA heteroduplex em cada cromossomo[2][3]. Essa estrutura pode ser vista na figura 2.
A molécula formada pela troca de fitas deve ser resolvida em duas moléculas duplex distintas, visto que as quatro fitas estão nesse momento da recombinação unidas como pode ser visto na figura 3. Essa configuração é chamada Junção de Holliday, uma estrutura ramificada de ácido nucleico que contém quatro braços de fita dupla unidos. Essas junções podem ser resolvidas com clivagens na mesma fita, resultando em recombinantes segmentados, apenas com um vestígio do evento de permuta, ou com clivagens nas outras fitas, resultando em cromossomos recombinantes. É possível ver a resolução de uma Junção de Holliday[4][5] também na figura 3 e 4.
Implicações da Recombinação Homóloga
editarA recombinação genética é de extrema importância para a evolução. Se não houvesse a possibilidade de permuta de material genético entre cromossomos homólogos, o conteúdo de cada cromossomo se tornaria fixo para seus alelos. Então quando mutações ocorressem, não seria possível separar as mutações favoráveis daquelas que são desfavoráveis. Assim, uma mutação desfavorável passaria a extensão do seu dano ao fitness do indivíduo de uma porção do genoma para todo o cromossomo em que ela se encontra[6]. Ao possibilitar a permuta entre os cromossomos através da recombinação, as mutações podem ser testadas como unidades individuais em novos arranjos pela combinação de alelos. Essa “mistura” permite a dissociação de alelos favoráveis aos desfavoráveis e sua disseminação na população.
O conhecimento do processo de recombinação também é importante para entender o desequilíbrio de ligação, uma vez que as taxas de recombinação podem aumentar ou diminuir a ligação de alelos no genoma. O desequilíbrio de ligação é importante na biologia evolutiva e na genética humana, porque muitos fatores o afetam e são afetados por ele. O desequilíbrio de ligação no genoma reflete a história da população, o sistema de reprodução e o padrão de subdivisão geográfica, enquanto o desequilíbrio de ligação em cada região genômica reflete o histórico de seleção natural, conversão gênica, mutação e outras forças que causam a evolução da frequência do gene. Como esses fatores afetam o desequilíbrio de ligação entre um par de loci específico ou em uma região genômica depende das taxas de recombinação local[7].
- ↑ a b c d Lewin, B. (LEWIN, Benjamin) (2009). Genes IX. Porto Alegre: Artmed. ISBN 9788536317540. OCLC 709271752
- ↑ Hunter, Neil; Kleckner, Nancy (julho de 2001). «The Single-End Invasion». Cell (em inglês). 106 (1): 59–70. doi:10.1016/S0092-8674(01)00430-5
- ↑ Szostak, Jack W.; Orr-Weaver, Terry L.; Rothstein, Rodney J.; Stahl, Franklin W. (maio de 1983). «The double-strand-break repair model for recombination». Cell. 33 (1): 25–35. ISSN 0092-8674. doi:10.1016/0092-8674(83)90331-8
- ↑ Lilley, David M. J.; White, Malcolm F. (junho de 2001). «The junction-resolving enzymes». Nature Reviews Molecular Cell Biology. 2 (6): 433–443. ISSN 1471-0072. doi:10.1038/35073057x
- ↑ West, Stephen C. (dezembro de 1997). «PROCESSING OF RECOMBINATION INTERMEDIATES BY THE RuvABC PROTEINS». Annual Review of Genetics. 31 (1): 213–244. ISSN 0066-4197. doi:10.1146/annurev.genet.31.1.213
- ↑ Lewin, B. (LEWIN, Benjamin) (2009). Genes IX. [S.l.]: Artmed. ISBN 9788536317540. OCLC 709271752
- ↑ Slatkin, Montgomery (junho de 2008). «Linkage disequilibrium — understanding the evolutionary past and mapping the medical future». Nature Reviews Genetics (em inglês). 9 (6): 477–485. ISSN 1471-0056. PMC 5124487
. PMID 18427557. doi:10.1038/nrg2361