ADN polimerase
| ADN polimerase ADN-dirixida | |||||||||
|---|---|---|---|---|---|---|---|---|---|

| |||||||||
| Estrutura tridimensional dos motivos hélice-xiro-hélice de unión ao ADN da ADN polimerase beta (baseado en PDB 7ICG ) | |||||||||
| Identificadores | |||||||||
| Número EC | 2.7.7.7 | ||||||||
| Número CAS | 9012-90-2 | ||||||||
| Bases de datos | |||||||||
| IntEnz | vista de IntEnz | ||||||||
| BRENDA | entrada de BRENDA | ||||||||
| ExPASy | vista de NiceZyme | ||||||||
| KEGG | entrada de KEGG | ||||||||
| MetaCyc | vía metabólica | ||||||||
| PRIAM | perfil | ||||||||
| Estruturas PDB | RCSB PDB PDBe PDBj PDBsum | ||||||||
| Gene Ontology | AmiGO / EGO | ||||||||
| |||||||||
Unha ADN polimerase ou ADN-pol (tamén DNA polimerase) é un encima que cataliza a polimerización de desoxirribonucleótidos formando unha cadea de ADN (replicación na cal axudan tamén outros encimas). A función esencial das ADN polimerases é realizar a replicación do ADN, na cal a polimerase "le" unha cadea de ADN que funciona como molde e sintetiza unha nova cadea complementaria. Por medio deste proceso cópiase unha molécula de ADN. A molécula acabada de polimerizar é complementaria en bases á cadea molde e, por tanto, idéntica á cadea orixinal unida á molde, que se separara dela para a replicación. As ADN polimerases utilizan ións magnesio como cofactores. As ADN polimerases humanas teñen unha lonxitude de 900 a 1000 aminoácidos.
Funcións
[editar | editar a fonte]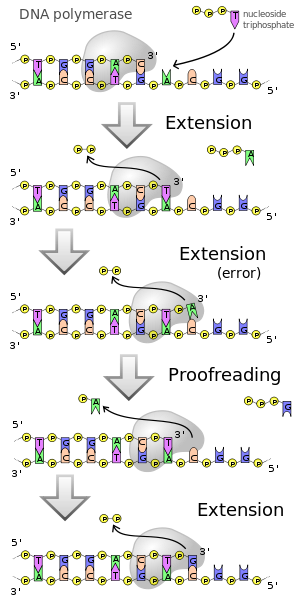
A ADN polimerase pode engadir nucleótidos libres só ao extremo 3' da cadea de ADN en formación. Isto fai que a elongación da cadea en formación se faga en dirección 5'-3'. Ningunha ADN polimerase coñecida pode empezar unha cadea de ADN de novo, é dicir, non pode colocar o primeiro nucleótido da cadea, despois unir o segundo, despois o terceiro etc. O único que poden facer é alongar unha cadea xa existente polo seu extremo 3'-OH, polo que sempre debe existir un fragmento inicial xa formado. Esta é a razón pola que a ADN polimerase necesita un cebador ou primer previamente formado ao cal engadir nucleótidos. Os cebadores ou primers poden estar formados por ARN ou ADN. Na replicación do ADN, as primeiras dúas bases son sempre de ARN, e son sintetizadas por outro encima chamado primase. Requírese tamén un encima chamado helicase para desenredar o ADN e desfacer nesa rexión a súa estrutura de dobre cadea e formar a estrutura de dúas cadeas simples bifurcadas (esta rexión é a forquita de replicación ou garfo de replicación), o cal facilita a replicación de cada unha das cadeas seguindo o modelo semiconservativo da replicación do ADN.
Outra propiedade que presentan algunhas ADN polimerases, pero non todas é a corrección de erros. Este proceso corrixe os erros producidos durante a formación do ADN neosintetizado. Cando se recoñece un par de bases colocado incorrectamente, a ADN polimerase inverte a dirección do seu movemento atrasándose un par de bases. Esta actividade de exonuclease 3'-5' do encima permite que o par de bases incorrecto sexa retirado para ser máis tarde substituído polo correcto pola propia polimerase, que continuará a replicación. Esta actividade denomínase corrección de probas.
As ADN polimerases son moi utilizadas nos experimentos de bioloxía molecular.
Variación entre especies
[editar | editar a fonte]As ADN polimerases teñen unha estrutura moi conservada, o cal significa que as súas subunidades catalíticas varían en conxunto moi pouco dunha especie a outra. As estruturas conservadas xeralmente exercen función importantes e insubstituíbles na célula, o mantemento das cales produce unha serie de vantaxes.
Algúns virus tamén codifican ADN polimerases especiais, como a ADN polimerase do virus da hepatite B, que replica especificamente ARN viral, e as transcriptases inversas dos retrovirus, as cales polimerizan ADN a partir dun molde de ARN.
Familias de ADN polimerases
[editar | editar a fonte]| ADN polimerase familia A | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 par c:o6-metil-guanina na polimerase | |||||||||
| Identificadores | |||||||||
| Símbolo | DNA_pol_A | ||||||||
| Pfam | PF00476 | ||||||||
| InterPro | IPR001098 | ||||||||
| PROSITE | PDOC00412 | ||||||||
| SCOPe | 1dpi / SUPFAM | ||||||||
| |||||||||
| ADN polimerase familia B | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 estrutura cristalina de rb69 gp43 formando un complexo con ADN que contén timina glicol | |||||||||
| Identificadores | |||||||||
| Símbolo | DNA_pol_B | ||||||||
| Pfam | PF00136 | ||||||||
| Pfam clan | CL0194 | ||||||||
| InterPro | IPR006134 | ||||||||
| PROSITE | PDOC00107 | ||||||||
| SCOPe | 1noy / SUPFAM | ||||||||
| |||||||||
| ADN polimerase tipo B, organular e viral | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 phi29 ADN polimerase, forma cristalina ortorrómbica, complexo ssDNA | |||||||||
| Identificadores | |||||||||
| Símbolo | DNA_pol_B_2 | ||||||||
| Pfam | PF03175 | ||||||||
| Pfam clan | CL0194 | ||||||||
| InterPro | IPR004868 | ||||||||
| |||||||||
Baseándose na homoloxía da súa secuencia de aminoácidos, as ADN polimerases poden ser subdivididas en sete familias: A, B, C, D, X, Y, e RT.
Familia A
[editar | editar a fonte]Esta familia de polimerases contén tanto polimerases replicativas coma reparadoras. Entre os membros replicativos da familia están a moi estudada T7 ADN polimerase, e a ADN polimerase mitocondrial eucariótica γ. As polimerases reparadoras son a ADN pol I de Escherichia coli, a pol I de Thermus aquaticus, e a pol I de Bacillus stearothermophilus. Estas polimerases reparadoras están implicadas na reparación de excisión e o procesamento de fragmentos de Okazaki xerados durante a síntese da fibra retardada ou descontinua na replicación do ADN.
Familia B
[editar | editar a fonte]As polimerases da familia B están implicadas na reparación e replicación do ADN. Entre elas están: Pol II (bacteriana), Pol B (arqueobacteriana), e Pol α, δ, e ε (eucarióticas). Outra polimerase B é a Pol ζ, feita de dúas subunidades, Rev3 e Rev7, que está implicada na síntese translesión. Pol ζ é única porque pode estender primers que teñen erros terminais sen complementariedade.
Familia C
[editar | editar a fonte]As polimerases C son os principais encimas replicativos cromosómicos bacterianos. A subunidade alfa da ADN polimerase III de E. coli é a subunidade catalítica [1] e non posúe actividade coñecida de nuclease. Outra subunidade, a epsilon, posúe actividade exonuclease 3'-5', que se utiliza para "editar" durante a replicación cromosómica. Agora clasifícanse ás veces as polimerases C como unha subcategoría da familia X.
Familia D
[editar | editar a fonte]Son polimerases aínda mal caracterizadas. Todos os exemplos coñecidos encóntranse no subdominio Euryarchaeota das Archaea e pénsase que son polimerasas replicativas.
Familia X
[editar | editar a fonte]Contén as ben coñecidas polimerases eucarióticas pol β, e outras polimerases eucarióticas como a pol σ, pol λ, pol μ, e a deoxinucleotidil transferase terminal (TdT). A Pol β requírese para a reparación de excisión de bases de remendo curto (short-patch, no que só se substitúe un nucleótido), unha vía de reparación do ADN que é esencial para a reparación de bases alquiladas e oxidadas e de sitios abásicos (onde falta unha base). A Pol λ e Pol μ están implicadas na unión de extremos non homólogos, un mecanismo para volver a unir as roturas no ADN de dobre cadea. A TdT exprésase só nos tecidos linfoides, e engade "n nucleótidos" ás roturas nunha dobre cadea formadas durante a recombinación dos xenes inmunitarios V(D)J para promover a diversidade inmunolóxica. O lévedo Saccharomyces cerevisiae ten só unha polimerase Pol X, a Pol IV, que está implicada na unión de extremos non homólogos.
Familia Y
[editar | editar a fonte]As polimerases Y difiren das outras en que teñen unha baixa fidelidade á hora de replicar ADNs molde non danados e na súa capacidade de replicar ADN danados, o que se coñece como síntese translesión (TLS). Os memberos desta familia denomínanse polimerases de síntese de translesión. Dependendo da lesión, as TLS polimerases poden saltar (by-pass) o dano dun modo libre de erros ou proclive aos erros, e este último dá lugar a unha elevada mutaxénese. Os pacientes de xeroderma pigmentoso variante (XPV) por exemplo, teñen mutacións na Pol η (eta) codificada nos seus xenes, que actúa no modo libre de erros para os danos por raios ultravioletas. Nos pacientes de XPV, as polimerases proclives a erros alternativos, por exemplo, a Pol ζ (zeta) (a polimerase ζ é unha polimerase da familia B, un complexo da subunidade catalítica REV3L con Rev7, que se asocia con Rev1[2]), pénsase que están implicadas en erros que orixinan a predisposición ao cáncer dos pacientes. Outros membros nos humanos son Pol ι (iota), Pol κ (kappa), e Rev1 (desoxicitidil transferase terminal). En E. coli coñécense dous TLS polimerases, a Pol IV (DINB) e a Pol V (UmuD'2C).
Familia RT
[editar | editar a fonte]Algúns virus tamén codifican ADN polimerases especiais, como a ADN polimerase do virus da hepatite B. Estas poden replicar selectivamente ADN viral de diversos modos. Os retrovirus codifican unha ADN polimerase especial chamada transcriptase inversa, que e unha ADN polimerase ARN dependente (RdDp). Polimeriza ADN a partir dun molde de ARN. A familia da transcriptase inversa contén exemplos de polimerases de retrovirus e de eucariotas. As polimerases eucarióticas están xeralmente restrinxidas a telomerases. Estas polimerases utilizan un molde de ARN para sintetizar a fibra de ADN.
ADN polimerases procarióticas
[editar | editar a fonte]Nas bacterias coñécense 5 ADN polimerases:
- Pol I: Implicada na reparación do ADN; ten unha actividade polimerase 5'→3', e actividades de exonuclease 3'→5' (corrección de probas) e 5'→3' (eliminación de primers de ARN).
- Pol II: Implicada na reparación de danos no ADNA; ten actividade de exonuclease 3'→5'.
- Pol III: A principal polimerase bacteriana (responsable da elongación da cadea); ten actividae de exonuclease 3'→5' (corrección de probas). Forma un complexo proteico de moitas subunidades chamado holoencima ADN polimerase III.
- Pol IV: Unha polimerase da famiilia Y; implicada na reparación SOS e na reparación translesión.
- Pol V: Unha polimerase da familia Y; participa no salto (by-pass) da zona danada; implicada na reparación SOS e na reparación translesión.
ADN polimerases eucarióticas
[editar | editar a fonte]Os eucariotas teñen polo menos 15 ADN polimerases.[3]
- POLA1, POLA2, PRIM1, PRIM2: Pol α: O complexo Pol α (complexo pol α-ADN primase) consta de catro subunidades: a subunidade catalítica POLA1, a subunidade reguladora POLA2, e as subunidades primase pequena e grande PRIM1 e PRIM2, respectivamente. Unha vez que a primase creou o cebador ou primer de ARN, a Pol α comeza a replicación elongando o cebador uns 20 nucleótidos. É membro da familia B de ADN polimerases.
- POLB: Pol β: Implicada na raparación do ADN, na reparación de excisión de bases e na síntese para encher zonas ausentes.
- POLG, POLG2: Pol γ: Replican e reparan o ADN mitocondrial e teñen unha actividade exonuclease 3'→5'.
- POLD1, POLD2, POLD3, POLD4: Pol δ: Moi activa na replicación e ademais con actividade exonuclease 3'→5' de corrección de probas. Pénsase que é a principal polimerase implicada na síntese da fibra retardada do ADN, pero aínda se debate o seu papel [4].
- POLE, POLE2, POLE3: Pol ε: Tamén moi activa na replicación e ademais con actividade exonuclease 3'→5' de corrección de probas. Moi relacionada con pol δ, e crese que é a principal polimerase implicada na síntese da fibra condutora ou guía do ADN [5], pero aínda se discute o seu papel [4].
- POLH, POLI, POLK, : η, ι, κ, e Rev1 son polimerases da familia Y e Pol ζ é da familia B. Estas polimerases están implicadas no salto (by-pass) da zona do ADN danada.[6]
- Coñécense tamén outras polimerases eucarióticas, que non foron ben caracterizadas aínda, que son:
Ningunha das polimerases eucarióticas pode eliminar os cabadores ou primers (actividade exonuclease 5'→3'); misión que cumpren outros encimas. Só as polimerases que fan a elongación (γ, δ e ε) teñen capacidade de corrección de probas (actividade exonuclease 3'→5').
Notas
[editar | editar a fonte]- ↑ Lamers, M.; Georgescu, R.; Lee, S.; O'Donnell, M.; Kuriyan, J. (2006). "Crystal structure of the catalytic alpha subunit of E. coli replicative DNA polymerase III". Cell 126 (5): 881–892. doi:10.1016/j.cell.2006.07.028. PMID 16959568.
- ↑ Gan GN; Wittschieben JP; Wittschieben BØ; Wood RD (2008). "DNA polymerase zeta (pol zeta) in higher eukaryotes". Cell Research 18 (1): 174–83. PMID 18157155. doi:10.1038/cr.2007.117.
- ↑ I. Hubscher, U.; Maga, G.; Spadari, S. (2002). "Eukaryotic DNA polymerases". Annual Review of Biochemistry 71: 133–63. PMID 12045093. doi:10.1146/annurev.biochem.71.090501.150041.
- ↑ 4,0 4,1 Scott D McCulloch; Thomas A Kunkel (01/2008). "The fidelity of DNA synthesis by eukaryotic replicative and translesion synthesis polymerases". Cell Research 18 (1): 148–161. PMID 18166979. doi:10.1038/cr.2008.4.
- ↑ Pursell, Z.F.; et al. (2007). "Yeast DNA Polymerase ε Participates in Leading-Strand DNA Replication". Science 317 (5834): 127–130. PMC 2233713. PMID 17615360. doi:10.1126/science.1144067.
- ↑ I. Prakash, S.; Johnson, R. E.; Prakash, L. (2005). "Eukaryotic translesion synthesis DNA polymerases: specificity of structure and function". Annual Review of Biochemistry 74: 317–53. PMID 15952890. doi:10.1146/annurev.biochem.74.082803.133250.
Véxase tamén
[editar | editar a fonte]Outros artigos
[editar | editar a fonte]Ligazóns externas
[editar | editar a fonte]- Burgers P; Koonin E; Bruford E; et al. (2001). "Eukaryotic DNA polymerases: proposal for a revised nomenclature". J. Biol. Chem. 276 (47): 43487–90. PMID 11579108. doi:10.1074/jbc.R100056200.
- PDB Molecule of the Month - pdb3_1Arquivado 18 de xullo de 2012 en Wayback Machine.
- Unusual repair mechanism in DNA polymerase lambda, Ohio State University
- MeshName - DNA+polymerases
- Número EC 2.7.7.7
- Animación do ADN Polimerase, WEHI, minuto 1:45 Arquivado 05 de decembro de 2014 en Wayback Machine.
| Wikimedia Commons ten máis contidos multimedia na categoría: ADN polimerase |